Seminario de Instrumentos Computacionales
R - Clase 3
Cristian Bonavida
Repaso Clase 2
En la clase 2, nos metimos de lleno a manipular datos. Aprendimos el ABC de dplyr para realizar las transformaciones esenciales que necesitamos al trabajar con datos tabulares
Repaso Clase 2
En la clase 2, nos metimos de lleno a manipular datos. Aprendimos el ABC de dplyr para realizar las transformaciones esenciales que necesitamos al trabajar con datos tabulares
Entre otras cosas aprendimos a:
filtrar observaciones
seleccionar y renombrar columnas
crear o modificar columnas
agrupar la base según alguna/s variables
generar tabla resumen
operar sobre múltiples columnas a la vez
Manipular bases
En esencia, estuvimos viendo cómo manipular filas (filtrar y generar subconjuntos de datos), cómo manipular columnas (transformarlas y generar nuevas) y cómo agrupar los datos para generar estimaciones o cálculos por grupo
Manipular bases
En esencia, estuvimos viendo cómo manipular filas (filtrar y generar subconjuntos de datos), cómo manipular columnas (transformarlas y generar nuevas) y cómo agrupar los datos para generar estimaciones o cálculos por grupo
Ahora veamos cómo manipular el dataframe como conjunto, para relacionarlo con otros
Manipular bases
En esencia, estuvimos viendo cómo manipular filas (filtrar y generar subconjuntos de datos), cómo manipular columnas (transformarlas y generar nuevas) y cómo agrupar los datos para generar estimaciones o cálculos por grupo
Ahora veamos cómo manipular el dataframe como conjunto, para relacionarlo con otros
Tipicamente existen dos tipos de operaciones entre bases de datos
Combinar bases de datos
Cruzar bases (Join)
Extender bases (Append)
Manipular bases
En esencia, estuvimos viendo cómo manipular filas (filtrar y generar subconjuntos de datos), cómo manipular columnas (transformarlas y generar nuevas) y cómo agrupar los datos para generar estimaciones o cálculos por grupo
Ahora veamos cómo manipular el dataframe como conjunto, para relacionarlo con otros
Tipicamente existen dos tipos de operaciones entre bases de datos
Combinar bases de datos
Cruzar bases (Join)
Extender bases (Append)
Reshape de una base de datos
Manipular bases
En esencia, estuvimos viendo cómo manipular filas (filtrar y generar subconjuntos de datos), cómo manipular columnas (transformarlas y generar nuevas) y cómo agrupar los datos para generar estimaciones o cálculos por grupo
Ahora veamos cómo manipular el dataframe como conjunto, para relacionarlo con otros
Tipicamente existen dos tipos de operaciones entre bases de datos
Combinar bases de datos
Cruzar bases (Join)
Extender bases (Append)
- Reshape de una base de datos
Manipular bases: join
El análisis de datos no implica trabajar, con una sola tabla o dataframe, mas bien se suele trabajar con múltiples bases y la mayoría de las veces es necesario combinarlos parar poder dar respuestas a las preguntas que motivan el análisis
Manipular bases: join
El análisis de datos no implica trabajar, con una sola tabla o dataframe, mas bien se suele trabajar con múltiples bases y la mayoría de las veces es necesario combinarlos parar poder dar respuestas a las preguntas que motivan el análisis
Varias tablas de datos se denominan datos relacionales por que para vincularlas es necesario establecer algún tipo de relación.
Manipular bases: join
El análisis de datos no implica trabajar, con una sola tabla o dataframe, mas bien se suele trabajar con múltiples bases y la mayoría de las veces es necesario combinarlos parar poder dar respuestas a las preguntas que motivan el análisis
Varias tablas de datos se denominan datos relacionales por que para vincularlas es necesario establecer algún tipo de relación.
Este trabajo relacional de combinar bases tiene como objetivo agregar nuevas variables a un marco de datos a partir de observaciones coincidentes en otro, lo que se conoce como mutating joins
Realizamos esta operación cuando necesitamos juntar columnas que están en dataframes diferentes
Manipular bases: join
El análisis de datos no implica trabajar, con una sola tabla o dataframe, mas bien se suele trabajar con múltiples bases y la mayoría de las veces es necesario combinarlos parar poder dar respuestas a las preguntas que motivan el análisis
Varias tablas de datos se denominan datos relacionales por que para vincularlas es necesario establecer algún tipo de relación.
Este trabajo relacional de combinar bases tiene como objetivo agregar nuevas variables a un marco de datos a partir de observaciones coincidentes en otro, lo que se conoce como mutating joins
Realizamos esta operación cuando necesitamos juntar columnas que están en dataframes diferentes
Por ejemplo, en la EPH individual tenemos columnas diferentes de las que tenemos en la EPH por hogar y (casi) siempre necesitamos juntarlas
Manipular bases: join
Para combinar dos bases necesitamos un atributo en común, es decir que haya una columna que se repita en ambas, que nos permita identificar a cada unidad en cada base para poder mantener las columnas de la base X y adosar las columnas de la base Y
Si pensamos en el caso de la base EPH individual y la EPH hogar tendríamos una estructura de este estilo:
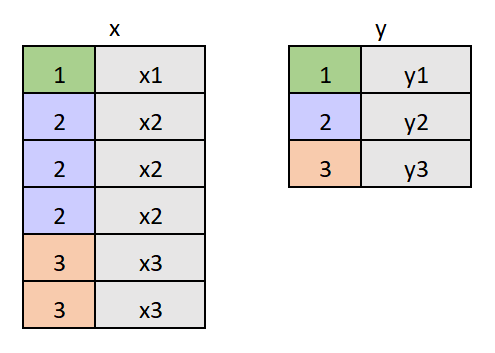
Manipular bases: join
En la base de individuos cada hogar se repite n veces, siendo n la cantidad de miembros del hogar.
Manipular bases: join
En la base de individuos cada hogar se repite n veces, siendo n la cantidad de miembros del hogar. En este caso el hogar 1 tiene un miembro, el hogar 2 tiene tres miembros y el hogar 3 tiene dos.
Manipular bases: join
En la base de individuos cada hogar se repite n veces, siendo n la cantidad de miembros del hogar. En este caso el hogar 1 tiene un miembro, el hogar 2 tiene tres miembros y el hogar 3 tiene dos.
Mientras que en la base de hogar tenemos una sola observación por hogar
Manipular bases: join
En la base de individuos cada hogar se repite n veces, siendo n la cantidad de miembros del hogar. En este caso el hogar 1 tiene un miembro, el hogar 2 tiene tres miembros y el hogar 3 tiene dos.
Mientras que en la base de hogar tenemos una sola observación por hogar
La estructura de las bases son distintas. Y entender cuál es la estructura de cada base en términos del identificador que las vincula, es importante para entender qué estamos haciendo.
Manipular bases: join
En la base de individuos cada hogar se repite n veces, siendo n la cantidad de miembros del hogar. En este caso el hogar 1 tiene un miembro, el hogar 2 tiene tres miembros y el hogar 3 tiene dos.
Mientras que en la base de hogar tenemos una sola observación por hogar
La estructura de las bases son distintas. Y entender cuál es la estructura de cada base en términos del identificador que las vincula, es importante para entender qué estamos haciendo.
Estamos combinando una base que tiene varias observaciones para un mismo identificador junto con otra que tiene una sola observación por identificador
Manipular bases: join
Según como sea la estructura de las bases a combinar tendremos 4 posibilidades diferentes:
- 1-1 (one to one)
Manipular bases: join
Según como sea la estructura de las bases a combinar tendremos 4 posibilidades diferentes:
- 1-1 (one to one) Ambas bases de datos tienen una sola observación por id
Manipular bases: join
Según como sea la estructura de las bases a combinar tendremos 4 posibilidades diferentes:
1-1 (one to one) Ambas bases de datos tienen una sola observación por id
1-m (one to many)
Manipular bases: join
Según como sea la estructura de las bases a combinar tendremos 4 posibilidades diferentes:
1-1 (one to one) Ambas bases de datos tienen una sola observación por id
1-m (one to many) La base de la izquierda tiene una única obs por id y a la derecha múltiples
Manipular bases: join
Según como sea la estructura de las bases a combinar tendremos 4 posibilidades diferentes:
1-1 (one to one) Ambas bases de datos tienen una sola observación por id
1-m (one to many) La base de la izquierda tiene una única obs por id y a la derecha múltiples
m-1 (many to one)
Manipular bases: join
Según como sea la estructura de las bases a combinar tendremos 4 posibilidades diferentes:
1-1 (one to one) Ambas bases de datos tienen una sola observación por id
1-m (one to many) La base de la izquierda tiene una única obs por id y a la derecha múltiples
m-1 (many to one) La base de la izquierda tiene múltiples obs por id y a la derecha una única
Manipular bases: join
Según como sea la estructura de las bases a combinar tendremos 4 posibilidades diferentes:
1-1 (one to one) Ambas bases de datos tienen una sola observación por id
1-m (one to many) La base de la izquierda tiene una única obs por id y a la derecha múltiples
m-1 (many to one) La base de la izquierda tiene múltiples obs por id y a la derecha una única
m-m (many to many)
Manipular bases: join
Según como sea la estructura de las bases a combinar tendremos 4 posibilidades diferentes:
1-1 (one to one) Ambas bases de datos tienen una sola observación por id
1-m (one to many) La base de la izquierda tiene una única obs por id y a la derecha múltiples
m-1 (many to one) La base de la izquierda tiene múltiples obs por id y a la derecha una única
m-m (many to many) Ambas bases tiene múltilpes observaaciones por id
Manipular bases: join
La estructura de la base resultante dependerá de la estructura de cada una
Manipular bases: join
La estructura de la base resultante dependerá de la estructura de cada una
Apliquemos el ejemplo de la PEH individual, a la cual queremos adosar algunas columnas de la EPH de hogares.
Manipular bases: join
La estructura de la base resultante dependerá de la estructura de cada una
Apliquemos el ejemplo de la PEH individual, a la cual queremos adosar algunas columnas de la EPH de hogares. Carguemos primero la base y seleccionemos 4 variables en cada una, aparte de las identificadoras
Manipular bases: join
La estructura de la base resultante dependerá de la estructura de cada una
Apliquemos el ejemplo de la PEH individual, a la cual queremos adosar algunas columnas de la EPH de hogares. Carguemos primero la base y seleccionemos 4 variables en cada una, aparte de las identificadoras
eph_ind <- read.csv("datos/Clase2/EPH_3T_22/usu_individual_T322.txt", sep=";", dec=",")eph_ind <- eph_ind %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, CH03, CH04, CH06) %>% rename_with(tolower) %>% rename(relacion=ch03, sexo=ch04, edad=ch06)eph_hog <- read.csv("datos/Clase2/EPH_3T_22/usu_hogar_T322.txt", sep=";", dec=",")eph_hog <- eph_hog %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, IV1, IV2, IV6, IV8) %>% rename_with(tolower) %>% rename(tipo_viv=iv1, n_ambiente=iv2, agua=iv6, baño=iv8)Manipular bases: join
La función inner_join() nos permite realizar esta combinación. Para ello debemos especificar 3 argumentos esenciales
x=dataframe de la izquierday=dataframe de la derechaby=variable/s identificadora
Manipular bases: join
La función inner_join() nos permite realizar esta combinación. Para ello debemos especificar 3 argumentos esenciales
x=dataframe de la izquierday=dataframe de la derechaby=variable/s identificadora
El atributo para vincular ambas bases será el identificador único que es la combinación de las variables CODUSU Y NRO_HOGAR,
eph_all <- inner_join(x=eph_ind, y=eph_hog, by=c("codusu", "nro_hogar"))Manipular bases: join
La función inner_join() nos permite realizar esta combinación. Para ello debemos especificar 3 argumentos esenciales
x=dataframe de la izquierday=dataframe de la derechaby=variable/s identificadora
El atributo para vincular ambas bases será el identificador único que es la combinación de las variables CODUSU Y NRO_HOGAR,
eph_all <- inner_join(x=eph_ind, y=eph_hog, by=c("codusu", "nro_hogar"))Revisemos la dimensión de la base resultante. Debería tener cuantas filas?...
Manipular bases: join
Pensemos en la estructura de los datos.
Manipular bases: join
Pensemos en la estructura de los datos. Realizamos una combinación del tipo m-1 (many to one)
Manipular bases: join
Pensemos en la estructura de los datos. Realizamos una combinación del tipo m-1 (many to one). Si logramos asignarle correctamente a cada miembro las variables de su hogar, deberíamos tener la misma cantidad de filas que la base individual
nrow(eph_all)==nrow(eph_ind)## [1] TRUEPero ahora nuestra base tiene el agregado de las columnas adosadas:
names(eph_all)## [1] "codusu" "nro_hogar" "aglomerado.x" "relacion" "sexo" ## [6] "edad" "aglomerado.y" "tipo_viv" "n_ambiente" "agua" ## [11] "baño"Manipular bases: join
Pensemos en la estructura de los datos. Realizamos una combinación del tipo m-1 (many to one). Si logramos asignarle correctamente a cada miembro las variables de su hogar, deberíamos tener la misma cantidad de filas que la base individual
nrow(eph_all)==nrow(eph_ind)## [1] TRUEPero ahora nuestra base tiene el agregado de las columnas adosadas:
names(eph_all)## [1] "codusu" "nro_hogar" "aglomerado.x" "relacion" "sexo" ## [6] "edad" "aglomerado.y" "tipo_viv" "n_ambiente" "agua" ## [11] "baño"En la base resultante tenemos para cada persona, las variables que corresponden a nivel hogar.
Manipular bases: join
Pensemos en la estructura de los datos. Realizamos una combinación del tipo m-1 (many to one). Si logramos asignarle correctamente a cada miembro las variables de su hogar, deberíamos tener la misma cantidad de filas que la base individual
nrow(eph_all)==nrow(eph_ind)## [1] TRUEPero ahora nuestra base tiene el agregado de las columnas adosadas:
names(eph_all)## [1] "codusu" "nro_hogar" "aglomerado.x" "relacion" "sexo" ## [6] "edad" "aglomerado.y" "tipo_viv" "n_ambiente" "agua" ## [11] "baño"En la base resultante tenemos para cada persona, las variables que corresponden a nivel hogar. Nuestro join fue exitoso!
Manipular bases: join
En el output de nombres vemos que R no duplicó las columnas que funcionaban de id, pero la columna de aglomerado que estaba presente en ambas bases quedó duplicada indicada con un subíndice por default.
Manipular bases: join
En el output de nombres vemos que R no duplicó las columnas que funcionaban de id, pero la columna de aglomerado que estaba presente en ambas bases quedó duplicada indicada con un subíndice por default.
A veces es útil mantener una misma columna de bases distintas, para chequear que el merge funcionó bien o simplemente porque necesitamos compararlas
Manipular bases: join
En el output de nombres vemos que R no duplicó las columnas que funcionaban de id, pero la columna de aglomerado que estaba presente en ambas bases quedó duplicada indicada con un subíndice por default.
A veces es útil mantener una misma columna de bases distintas, para chequear que el merge funcionó bien o simplemente porque necesitamos compararlas . En estos casos el argumento suffix= nos permite agregar un sufijo a elección, por ejemplo:
eph_all <- inner_join(x=eph_ind, y=eph_hog, by=c("codusu", "nro_hogar"), suffix = c("_ind", "_hog"))names(eph_all)## [1] "codusu" "nro_hogar" "aglomerado_ind" "relacion" ## [5] "sexo" "edad" "aglomerado_hog" "tipo_viv" ## [9] "n_ambiente" "agua" "baño"Manipular bases: join
El ejemplo con la EPH es una caso particular donde por construcción todas las observaciones en la base de individuos encuentran una correspondencia con el id por hogar
Manipular bases: join
El ejemplo con la EPH es una caso particular donde por construcción todas las observaciones en la base de individuos encuentran una correspondencia con el id por hogar
En un mismo trimestre nunca tendremos el caso de que en la base de la izquierda hay observaciones que no matcheen con al menos una de la derecha, ni viceversa
Manipular bases: join
El ejemplo con la EPH es una caso particular donde por construcción todas las observaciones en la base de individuos encuentran una correspondencia con el id por hogar
En un mismo trimestre nunca tendremos el caso de que en la base de la izquierda hay observaciones que no matcheen con al menos una de la derecha, ni viceversa
Sin embargo esto sí es algo muy común al trabajar con cualquier otra bases de datos. Es probable que al combinar dos dataframes nos encontremos con un caso como este o similar:
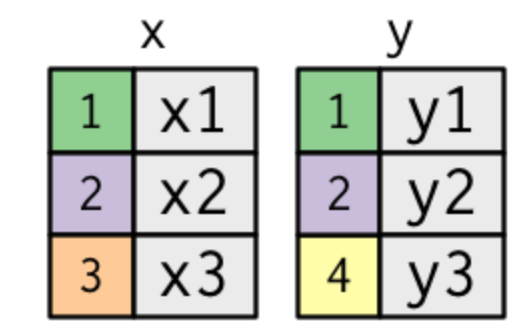
Manipular bases: join
Pero qué pasa si tenemos observaciones diferentes en cada base.
Manipular bases: join
Pero qué pasa si tenemos observaciones diferentes en cada base. Es decir en la base X tenemos filas que no están presentes en la base Y, o viceversa, o ambos casos en simultaneo
Manipular bases: join
Pero qué pasa si tenemos observaciones diferentes en cada base. Es decir en la base X tenemos filas que no están presentes en la base Y, o viceversa, o ambos casos en simultaneo
¿Queremos mantener todas, solo las de la base X, o solo las de la base Y?
Manipular bases: join
Pero qué pasa si tenemos observaciones diferentes en cada base. Es decir en la base X tenemos filas que no están presentes en la base Y, o viceversa, o ambos casos en simultaneo
¿Queremos mantener todas, solo las de la base X, o solo las de la base Y?
Cómo combinamos o preservamos las filas entre bases depende del tipo de cruce que haremos. Es decir del criterio con el que definimos este emparejamiento o match entre filas.
Manipular bases: join
Pero qué pasa si tenemos observaciones diferentes en cada base. Es decir en la base X tenemos filas que no están presentes en la base Y, o viceversa, o ambos casos en simultaneo
¿Queremos mantener todas, solo las de la base X, o solo las de la base Y?
Cómo combinamos o preservamos las filas entre bases depende del tipo de cruce que haremos. Es decir del criterio con el que definimos este emparejamiento o match entre filas.
En el ejemplo anterior utilizamos el inner_join(), pero en realidad este es una de 4 las opciones que podemos emplear para definir un match
- Inner join mantiene solo las observaciones comunes entre x e y
- Left join mantiene todas las observaciones que están en x
- Right join mantiene todas las observaciones que están en y
- Full join mantiene todas las observaciones que están tanto en x como en y
Manipular bases: join
En la figura anterior ilustramos un caso donde las observaciones difieren entre bases, incluso cuando tengamos una relación de 1-1 (one to one)
Manipular bases: join
En la figura anterior ilustramos un caso donde las observaciones difieren entre bases, incluso cuando tengamos una relación de 1-1 (one to one)
La figura nos ayuda a clarificar que, el tipo de relación o correspondencia entre observaciones y los casos de observaciones faltantes entre bases, aunque puedan confundirse, son en realidad son dos cuestinoes diferentes
- El tipo de relación o correspondencia: refiere a si un mismo id matchea con mas de una fila en alguna de las bases
Manipular bases: join
En la figura anterior ilustramos un caso donde las observaciones difieren entre bases, incluso cuando tengamos una relación de 1-1 (one to one)
La figura nos ayuda a clarificar que, el tipo de relación o correspondencia entre observaciones y los casos de observaciones faltantes entre bases, aunque puedan confundirse, son en realidad son dos cuestinoes diferentes
El tipo de relación o correspondencia: refiere a si un mismo id matchea con mas de una fila en alguna de las bases
Las observaciones faltantes o diferentes: responde al criterio de qué hacer con las filas que no encuentran un match
Manipular bases: join
En la figura anterior ilustramos un caso donde las observaciones difieren entre bases, incluso cuando tengamos una relación de 1-1 (one to one)
La figura nos ayuda a clarificar que, el tipo de relación o correspondencia entre observaciones y los casos de observaciones faltantes entre bases, aunque puedan confundirse, son en realidad son dos cuestinoes diferentes
El tipo de relación o correspondencia: refiere a si un mismo id matchea con mas de una fila en alguna de las bases
Las observaciones faltantes o diferentes: responde al criterio de qué hacer con las filas que no encuentran un match
Manipular bases: join
Más allá del tipo de relación entre bases (1-1; m-1; 1-m; m-m) podemos tener filas que no encuentren un match
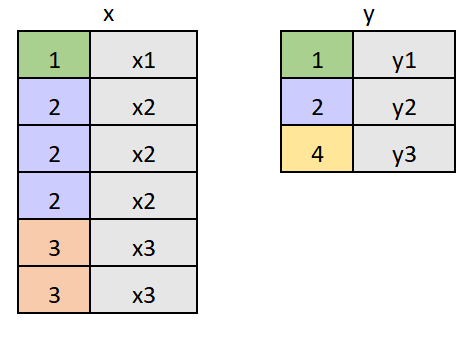
Manipular bases: join
Más allá del tipo de relación entre bases (1-1; m-1; 1-m; m-m) podemos tener filas que no encuentren un match

Entonces debemos definir un criterio para tratar estas filas sin match, es decir para preservarlas o descartarlas según en qué base se encuentren. Esto es lo que hacemos con laos 4 tipos de funciones join: inner_join() left_join() right_join() full_join()
Manipular bases: join
Más allá del tipo de relación entre bases (1-1; m-1; 1-m; m-m) podemos tener filas que no encuentren un match

Entonces debemos definir un criterio para tratar estas filas sin match, es decir para preservarlas o descartarlas según en qué base se encuentren. Esto es lo que hacemos con laos 4 tipos de funciones join: inner_join() left_join() right_join() full_join()
Veamos cada una en detalle. Para ilustrar usaremos la relación mas simple de 1-1 y haremos la aplicación con un sampleo de las bases de EPH, por lo que la relación sera de m-1
Manipular bases: inner_join
Comencemos con esta función que ya empleamos antes.
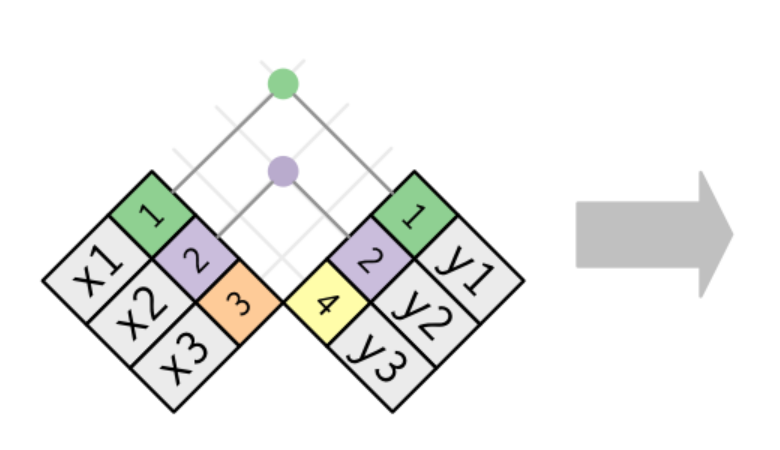
Manipular bases: inner_join
Este es el caso mas estricto, donde solo mantenemos las filas que encuentran un match
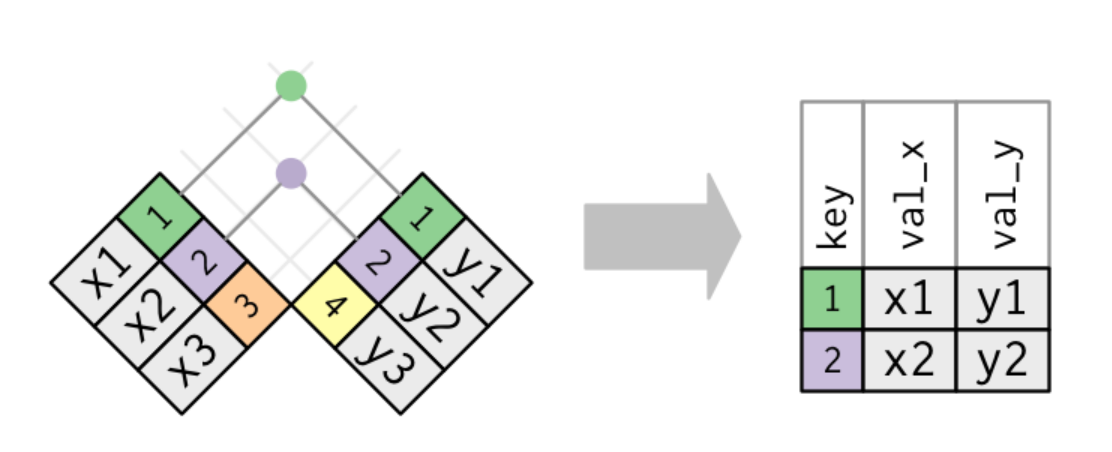
Manipular bases: inner_join
Para aplicar el caso, como la EPH por construcción no presenta id faltantes en las bases, generamos un ejemplo artificial con un sampleo aleatorio del 75% la base de individuos y de hogares.
Manipular bases: inner_join
Para aplicar el caso, como la EPH por construcción no presenta id faltantes en las bases, generamos un ejemplo artificial con un sampleo aleatorio del 75% la base de individuos y de hogares.
- base X: random sample de la base de individuos
- base y: random sample de la base de hogares
Manipular bases: inner_join
Para aplicar el caso, como la EPH por construcción no presenta id faltantes en las bases, generamos un ejemplo artificial con un sampleo aleatorio del 75% la base de individuos y de hogares.
- base X: random sample de la base de individuos
- base y: random sample de la base de hogares
eph_ind_samp <- sample_frac(eph_ind, 0.75, replace = FALSE)eph_hog_samp <- sample_frac(eph_hog, 0.75, replace = FALSE)De esta forma tendremos algunas observaciones faltantes en cada base
Manipular bases: inner_join
La función en este caso es inner_join() y los argumentos a especificar son los mismos que antes
x=dataframe de la izquierday=dataframe de la derechaby=variable/s identificadora
comunes <- inner_join(eph_ind_samp, eph_hog_samp, by=c("codusu", "nro_hogar"))Manipular bases: inner_join
La funcion setdiff() nos devuelve las filas que aparecen en un vector pero no en otro. De esta forma, si resumimos "codusu" y "nro_hogar" en una sola columna "id" podemos testear facilmente nuestro resultado
eph_hog_samp$id <- paste(eph_hog_samp$codusu, eph_hog_samp$nro_hogar, sep="-")eph_ind_samp$id <- paste(eph_ind_samp$codusu, eph_ind_samp$nro_hogar, sep="-") #Rows that appear in "ind" but not "hog"out_izq <- setdiff(eph_ind_samp$id, eph_hog_samp$id)#Rows that appear in "hog" but not "ind"out_der <- setdiff(eph_hog_samp$id, eph_ind_samp$id)#Check si los id en el inner_join están en las lista de los "out"comunes$id <- paste(comunes$codusu, comunes$nro_hogar, sep="-") nrow( comunes %>% filter(id %in% out_izq | id %in% out_der) )## [1] 0Vemos que ninguno de los "id" que no encuentran match entre bases, están incluidos en el dataframe resultante del inner_join
Manipular bases: left_join
En este segundo criterio mantendremos siempre las filas presentes en la base x
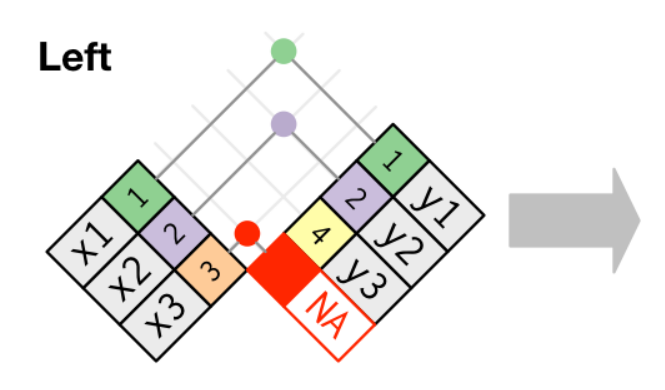
Manipular bases: left_join
En este segundo criterio mantendremos siempre las filas presentes en la base x
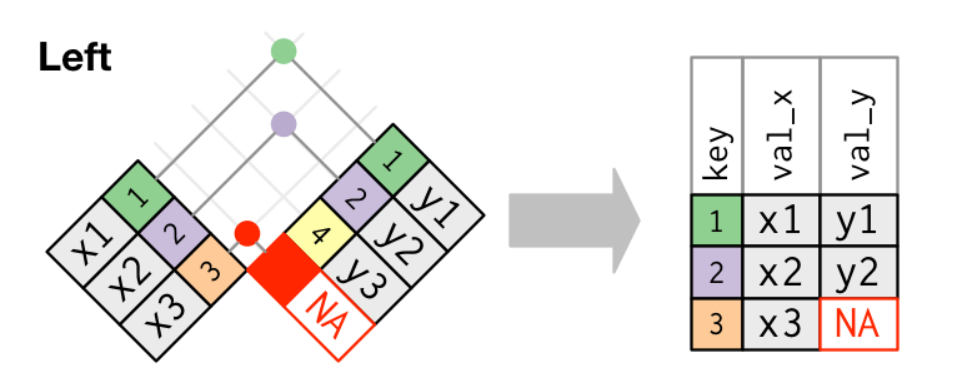
Manipular bases: left_join
Como pueden intuir la función es left_join() y los argumentos a especificar son los mismos que antes
todas_x <- left_join(eph_ind_samp, eph_hog_samp, by=c("codusu", "nro_hogar"))Manipular bases: left_join
Como pueden intuir la función es left_join() y los argumentos a especificar son los mismos que antes
todas_x <- left_join(eph_ind_samp, eph_hog_samp, by=c("codusu", "nro_hogar"))La cantidad de filas de la base resultante tiene que ser la misma que la base indicada como x
nrow(todas_x)==nrow(eph_ind_samp)## [1] TRUEManipular bases: left_join
Como pueden intuir la función es left_join() y los argumentos a especificar son los mismos que antes
todas_x <- left_join(eph_ind_samp, eph_hog_samp, by=c("codusu", "nro_hogar"))La cantidad de filas de la base resultante tiene que ser la misma que la base indicada como x
nrow(todas_x)==nrow(eph_ind_samp)## [1] TRUEPero la cantidad de columnas ser mayor,
dim(todas_x)## [1] 36924 13Manipular bases: right_join
Bajo este criterio mantendremos siempre las filas presentes en la base y
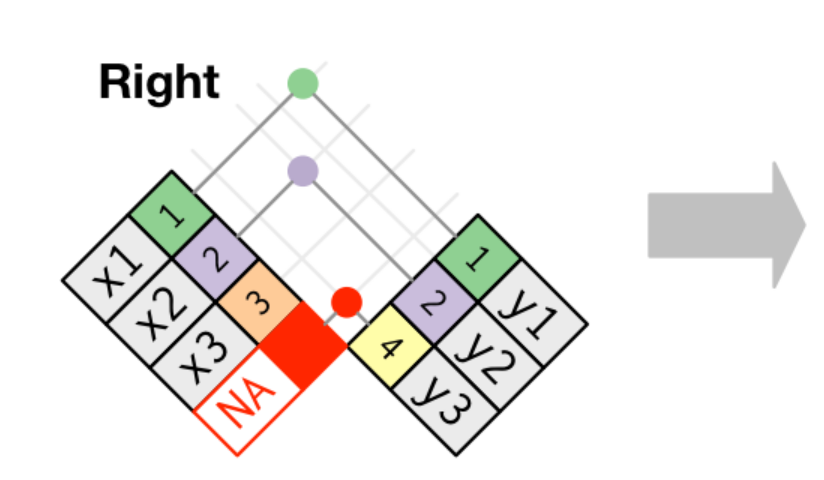
Manipular bases: right_join
Bajo este criterio mantendremos siempre las filas presentes en la base y
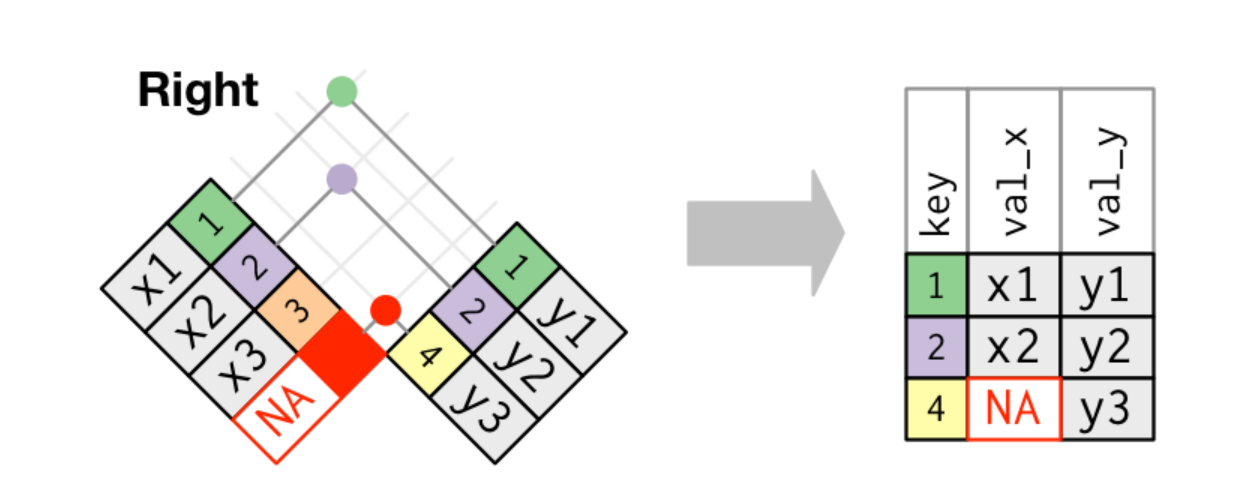
Manipular bases: right_join
La función a emplear es right_join()
todas_y <- right_join(eph_ind_samp, eph_hog_samp, by=c("codusu", "nro_hogar"))La cantidad de filas de la base resultantes tiene que ser la misma que en la base indicada como y?
Manipular bases: right_join
La función a emplear es right_join()
todas_y <- right_join(eph_ind_samp, eph_hog_samp, by=c("codusu", "nro_hogar"))La cantidad de filas de la base resultantes tiene que ser la misma que en la base indicada como y?
nrow(todas_y)==nrow(eph_hog_samp)## [1] FALSEManipular bases: right_join
La función a emplear es right_join()
todas_y <- right_join(eph_ind_samp, eph_hog_samp, by=c("codusu", "nro_hogar"))La cantidad de filas de la base resultantes tiene que ser la misma que en la base indicada como y?
nrow(todas_y)==nrow(eph_hog_samp)## [1] FALSELa cantidad de filas no duplicadas de la base resultantes tiene que ser la misma que en la base indicada como y
Manipular bases: right_join
La función a emplear es right_join()
todas_y <- right_join(eph_ind_samp, eph_hog_samp, by=c("codusu", "nro_hogar"))La cantidad de filas de la base resultantes tiene que ser la misma que en la base indicada como y?
nrow(todas_y)==nrow(eph_hog_samp)## [1] FALSELa cantidad de filas no duplicadas de la base resultantes tiene que ser la misma que en la base indicada como y. Y la cantidad de columnas tiene que ser mayor,
nrow(distinct(todas_y, codusu, nro_hogar))==nrow(eph_hog_samp)dim(todas_y)## [1] TRUE## [1] 28610 13Manipular bases: full_join
Este criterio es el mas abarcativo de todos, ya que considera todas las filas
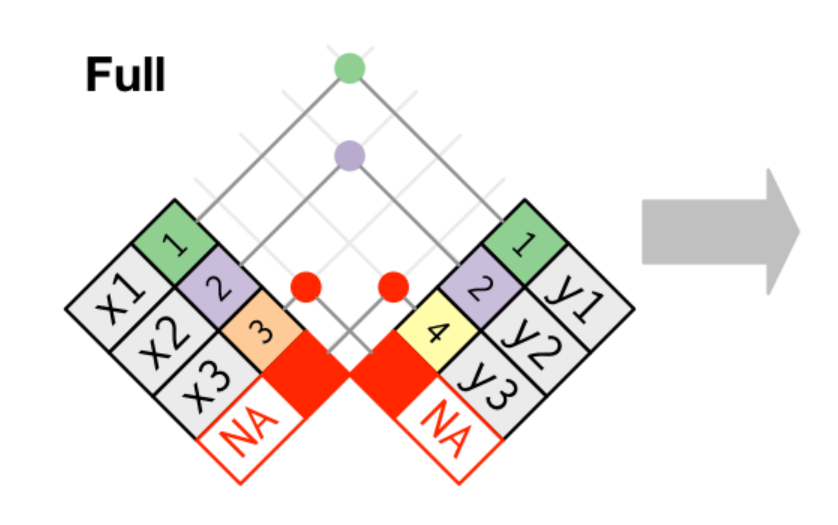
Manipular bases: full_join
Este criterio es el mas abarcativo de todos, ya que considera todas las filas
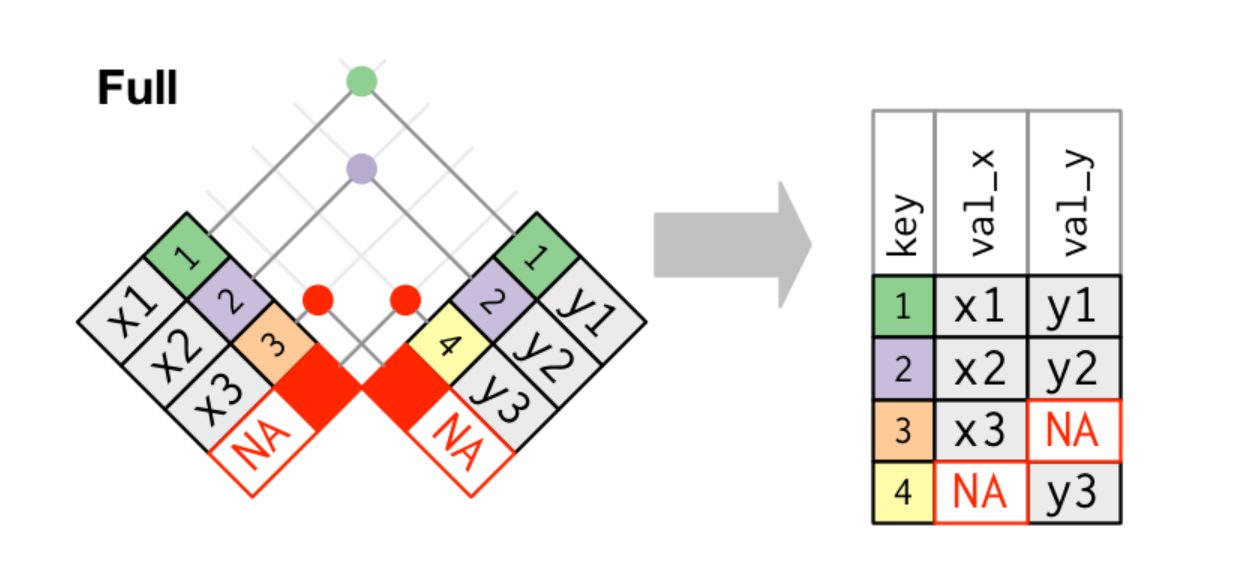
Manipular bases: full_join
La función en este caso es full_join()
todas <- full_join(eph_ind_samp, eph_hog_samp, by=c("codusu", "nro_hogar"))Chequemos que esta df sea el conjunto mas amplio de todos los joins anteriores y que contenga tanto a los id faltantes de la base de individuos como los de hogares
Manipular bases: full_join
La función en este caso es full_join()
todas <- full_join(eph_ind_samp, eph_hog_samp, by=c("codusu", "nro_hogar"))Chequemos que esta df sea el conjunto mas amplio de todos los joins anteriores y que contenga tanto a los id faltantes de la base de individuos como los de hogares
nrow(todas)>nrow(comunes)nrow(todas)>nrow(todas_y)nrow(todas)>nrow(todas_x)## [1] TRUE## [1] TRUE## [1] TRUEManipular bases: full_join
La función en este caso es full_join()
todas <- full_join(eph_ind_samp, eph_hog_samp, by=c("codusu", "nro_hogar"))Chequemos que esta df sea el conjunto mas amplio de todos los joins anteriores y que contenga tanto a los id faltantes de la base de individuos como los de hogares
nrow(todas)>nrow(comunes)nrow(todas)>nrow(todas_y)nrow(todas)>nrow(todas_x)## [1] TRUE## [1] TRUE## [1] TRUE#Check si los id sin correspondencia están incluidos en la base resultantetodas$id <- paste(todas$codusu, todas$nro_hogar, sep="-") nrow( todas %>% filter(id %in% out_izq | id %in% out_der) )## [1] 10268Manipular bases: tips for join
Antes de pasar al tema siguiente, recomiendo poder examinar otros ejemplos y aplicaciones de joins mas allá de este ejemplo que hemos presentado con la EPH.
Manipular bases: tips for join
Antes de pasar al tema siguiente, recomiendo poder examinar otros ejemplos y aplicaciones de joins mas allá de este ejemplo que hemos presentado con la EPH.
Este tipo de operaciones es de las operaciones mas frecuentes al trabajar con datos y son fuente de errores o modificaciones no deseadas en los datos
Manipular bases: tips for join
Antes de pasar al tema siguiente, recomiendo poder examinar otros ejemplos y aplicaciones de joins mas allá de este ejemplo que hemos presentado con la EPH.
Este tipo de operaciones es de las operaciones mas frecuentes al trabajar con datos y son fuente de errores o modificaciones no deseadas en los datos
Siempre es recomendable tomarse unos minutos para analizar lo siguiente antes de un join:
- Averiguar cómo es la correspondencia entre las observaciones de cada base? 1-1; m-1; 1-m?
- Evitar cruces que implique relaciones m-m, la base resultante es difícil de interpretar y rara vez es necesario este tipo de combinaciones
- Averiguar si tengo filas diferentes en cada base? Siempre es bueno realizar un check de filas faltantes con
setdiff() - Entender cuál es la estructura del dataframe resultante que necesito
- En base a ellos pensar que criterio de join se debe emplear
- Lo mas importante, chequear que el resultado sea el correcto y no darlo por hecho
Manipular bases: errores mas comunes en join
Manipular bases: append
Pasemos ahora a otro tipo de operaciones, que implicar combinar bases, pero no matchear sino que juntatarlas
Tipicamente existen dos tipos de operaciones entre bases
Combinar bases de datos
Cruzar bases (Join)
Extender bases (Append)
- Reshape de una base de datos
Manipular bases: append
Cuando tenemos dos o mas bases de datos y queremos juntarlas en una sola, lo que estamos buscando es extender la base, es decir sumar las filas de bases diferentes
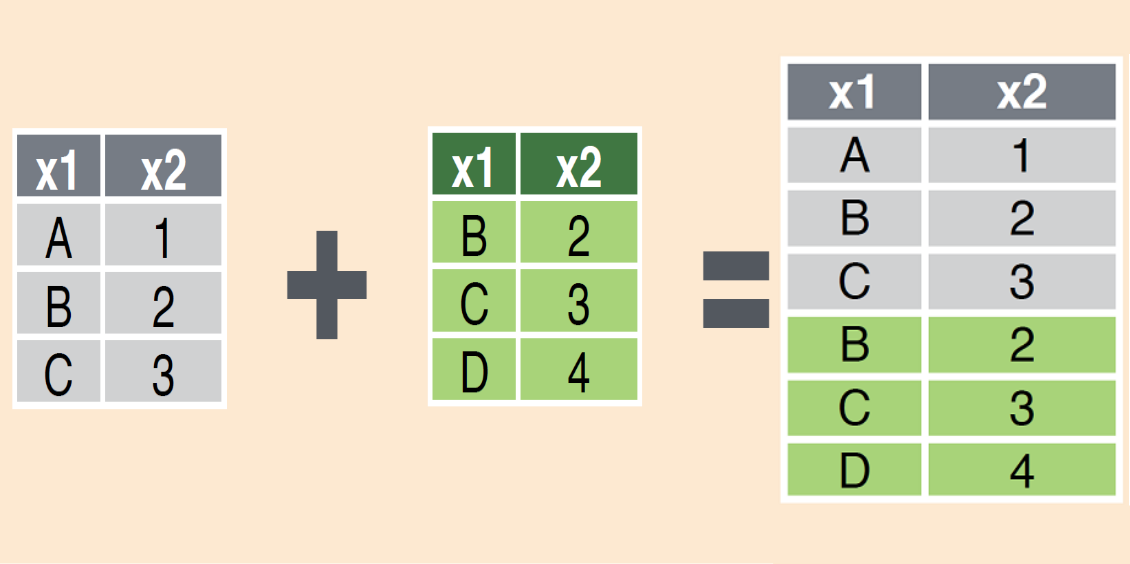
Manipular bases: append
Cuando tenemos dos o mas bases de datos y queremos juntarlas en una sola, lo que estamos buscando es extender la base, es decir sumar las filas de bases diferentes

Por lo general, y en la mayoría de los casos estas bases comparten las mismas columnas y lo que precisamos es apilarlas
Manipular bases: append
Cuando tenemos dos o mas bases de datos y queremos juntarlas en una sola, lo que estamos buscando es extender la base, es decir sumar las filas de bases diferentes

Por lo general, y en la mayoría de los casos estas bases comparten las mismas columnas y lo que precisamos es apilarlas
Siguiendo con la EPH el ejemplo más tipico, ocurre cuando para una base de individuos u hogares de un trimestre, queremos sumarle la misma base de un trimestre anterior
Manipular bases: append
Veamos cómo hacer esto en R.
Manipular bases: append
Veamos cómo hacer esto en R. La función para este tipo de operaciones como su nombre lo indica es bind_rows()
Manipular bases: append
Veamos cómo hacer esto en R. La función para este tipo de operaciones como su nombre lo indica es bind_rows(). Y requiere solo dos argumentos, los dataframes que queremos juntar
Manipular bases: append
Veamos cómo hacer esto en R. La función para este tipo de operaciones como su nombre lo indica es bind_rows(). Y requiere solo dos argumentos, los dataframes que queremos juntar
Para nuestra aplicación, a la base de individuos del 3er trimestre 2022 con las columnas elegidas antes, agreguemosle la base del 2do trimestre
Manipular bases: append
Veamos cómo hacer esto en R. La función para este tipo de operaciones como su nombre lo indica es bind_rows(). Y requiere solo dos argumentos, los dataframes que queremos juntar
Para nuestra aplicación, a la base de individuos del 3er trimestre 2022 con las columnas elegidas antes, agreguemosle la base del 2do trimestre
Primero carguemos la base y renombramos la EPH individual que ya estaba cargada en nuestro entorno de trabajo
eph_ind_2t <- read.csv("datos/Clase3/EPH_2T_22/usu_individual_T222.txt", sep=";", dec=",")eph_ind_2t <- eph_ind_2t %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, CH03, CH04, CH06) %>% rename_with(tolower) %>% rename(relacion=ch03, sexo=ch04, edad=ch06)eph_ind_3t <- eph_indManipular bases: append
Ya estamos listos para hacer el append
eph_ind_append <- bind_rows(eph_ind_2t, eph_ind_3t)Corroboremos que la cantidad de filas corresponden a la suma de cada base
nrow(eph_ind_append)==( nrow(eph_ind_2t) + nrow(eph_ind_3t) )## [1] TRUEListo!
Manipular bases: append
¿Y que ocurre en el caso de que las columnas que tenemos en cada base sean distintas y queramos apilar las bases sin perder las columnas propias de cada base?
Bueno, en realidad no pasa nada, la función puede lidiar con estos casos
Manipular bases: append
¿Y que ocurre en el caso de que las columnas que tenemos en cada base sean distintas y queramos apilar las bases sin perder las columnas propias de cada base?
Bueno, en realidad no pasa nada, la función puede lidiar con estos casos
Cuando se hace un append de filas, las columnas se emparejan por nombre y las columnas faltantes se completarán con NA
eph_ind_3t <- eph_ind_3t %>% select(-edad) eph_ind_2t <- eph_ind_2t %>% select(-sexo)eph_ind_append <- bind_rows(eph_ind_2t, eph_ind_3t)Manipular bases: append
Con head() veremos las primeras observaciones que son las del 2do-trim y con tail() nos devuelve las ultimas observaciones, que corresponden a las filas del 3er-trim, en ambos casos con NA en la variable faltante
head(eph_ind_append, 5)## codusu nro_hogar aglomerado relacion edad sexo## 1 TQRMNOPWWHMMLNCDEFPCH00700147 1 9 1 50 NA## 2 TQRMNOPXVHLMLMCDEIJAH00706342 1 33 1 52 NA## 3 TQRMNOPXVHLMLMCDEIJAH00706342 1 33 2 45 NA## 4 TQRMNOPXVHLMLMCDEIJAH00706342 1 33 3 21 NA## 5 TQRMNOPXVHLMLMCDEIJAH00706342 1 33 3 12 NAtail(eph_ind_append, 5)## codusu nro_hogar aglomerado relacion edad sexo## 99842 TQRMNOQTYHLNKPCDEIHJF00707321 1 31 1 NA 1## 99843 TQRMNOSVTHLNMQCDEIJAH00785774 1 33 3 NA 1## 99844 TQRMNOSVTHLNMQCDEIJAH00785774 1 33 3 NA 2## 99845 TQRMNOSVTHLNMQCDEIJAH00785774 1 33 1 NA 2## 99846 TQRMNOSVTHLNMQCDEIJAH00785774 1 33 2 NA 1Manipular bases: append
Un tip adicional sobre bind_rows()
- Pueden explorar el argumento adicional
id, que indica que se cree una columna nueva para identifcar de qué dataframe provino cada observación.
Manipular bases: append
Un tip adicional sobre bind_rows()
- Pueden explorar el argumento adicional
id, que indica que se cree una columna nueva para identifcar de qué dataframe provino cada observación. Esto en ciertos contextos resulta muy útil
Manipular bases: append
Un tip adicional sobre bind_rows()
- Pueden explorar el argumento adicional
id, que indica que se cree una columna nueva para identifcar de qué dataframe provino cada observación. Esto en ciertos contextos resulta muy útil
eph_ind_append <- bind_rows(eph_ind_2t, eph_ind_3t, .id = "origen")table(eph_ind_append$origen)## ## 1 2 ## 50614 49232Manipular bases: append
Existe también la función bind_cols() que permite juntar las columnas de bases distintas.
Manipular bases: append
Existe también la función bind_cols() que permite juntar las columnas de bases distintas.
Sin embargo deben ser cuidadosos a la hora de usarla ya que al juntar columnas se toma en cuenta la posición de las filas. Solo es recomendable cuando están seguros de que las observaciones son las mismas y están ordenadas con la misma posición en cada base de datos
Manipular bases: append
Existe también la función bind_cols() que permite juntar las columnas de bases distintas.
Sin embargo deben ser cuidadosos a la hora de usarla ya que al juntar columnas se toma en cuenta la posición de las filas. Solo es recomendable cuando están seguros de que las observaciones son las mismas y están ordenadas con la misma posición en cada base de datos
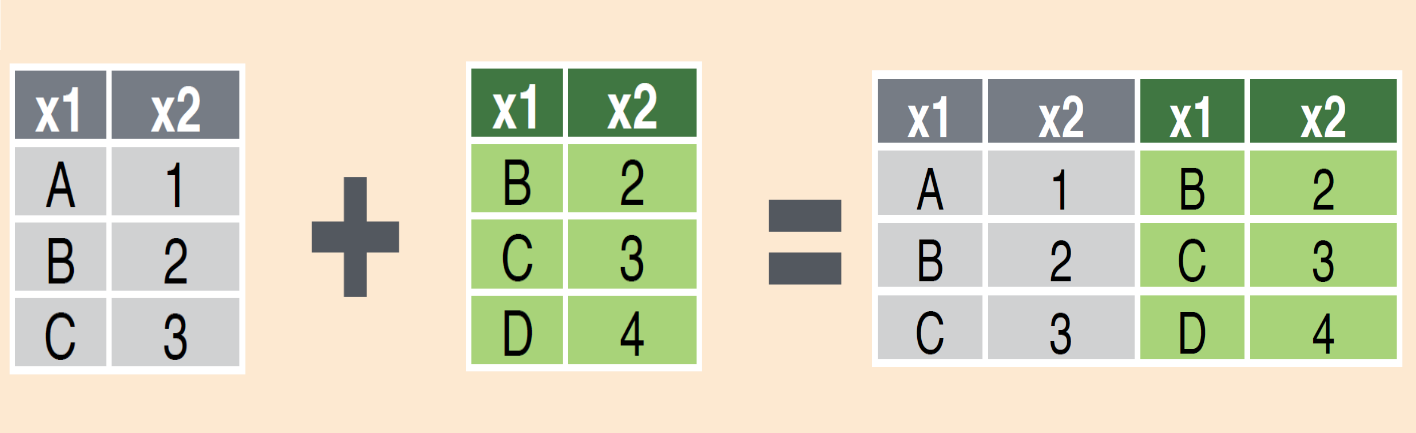
Manipular bases: append
Existe también la función bind_cols() que permite juntar las columnas de bases distintas.
Sin embargo deben ser cuidadosos a la hora de usarla ya que al juntar columnas se toma en cuenta la posición de las filas. Solo es recomendable cuando están seguros de que las observaciones son las mismas y están ordenadas con la misma posición en cada base de datos
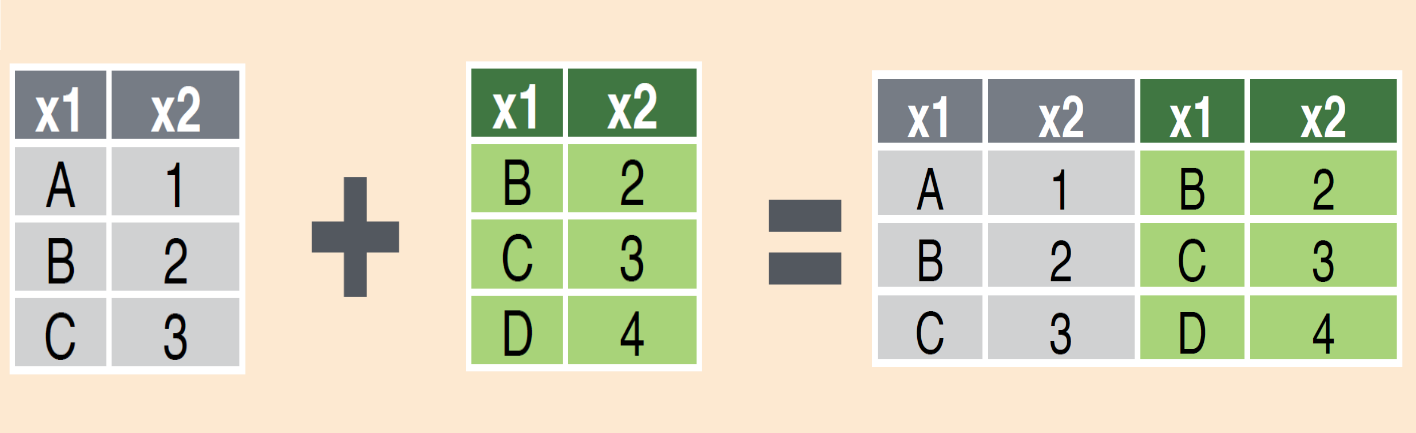
En caso contrario, y para evitar asiganciones incorrectas, es mejor usar los mutating joins!
Manipular bases: pivot
Pasemos a otro tipo de operaciones que se dan cuando necesitamos cambiar la estructura de la base.
Combinar bases de datos
Cruzar bases (Join)
Extender bases (Append)
- Reshape de una base de datos
Manipular bases: pivot
Un mismo dataframe puede almacenar los mismos datos, pero en estructuras distintas.
Realizar un reshape o pivotar una base de datos, implica mantener tal cual los valores y la información de la tabla, alterando la forma o la disposición de esos datos:
Manipular bases: pivot
Un mismo dataframe puede almacenar los mismos datos, pero en estructuras distintas.
Realizar un reshape o pivotar una base de datos, implica mantener tal cual los valores y la información de la tabla, alterando la forma o la disposición de esos datos:
Existen dos formatos para una dataframe:
- long: en el cual los datos estan apilados, una misma observación se repite ya que los distintos valores de la columna se ordenan uno debajo del otro
Manipular bases: pivot
Un mismo dataframe puede almacenar los mismos datos, pero en estructuras distintas.
Realizar un reshape o pivotar una base de datos, implica mantener tal cual los valores y la información de la tabla, alterando la forma o la disposición de esos datos:
Existen dos formatos para una dataframe:
long: en el cual los datos estan apilados, una misma observación se repite ya que los distintos valores de la columna se ordenan uno debajo del otro
wide: los datos están dispuestos en sentido horizontal, con múltiples columnas y con observaciones que no se repiten
Manipular bases: pivot
Para ver un caso, trabajemos con una base ejemplo que tiene para cada región los distintos valores del ingreso laboral en distintos años:
inc_reg <- read.csv("datos/Clase3/inc_region.csv")Visualicemos nuestra base
Manipular bases: pivot
Para ver un caso, trabajemos con una base ejemplo que tiene para cada región los distintos valores del ingreso laboral en distintos años:
inc_reg <- read.csv("datos/Clase3/inc_region.csv")Visualicemos nuestra base Qué formato tiene
Manipular bases: pivot
Para ver un caso, trabajemos con una base ejemplo que tiene para cada región los distintos valores del ingreso laboral en distintos años:
inc_reg <- read.csv("datos/Clase3/inc_region.csv")Visualicemos nuestra base Qué formato tiene ¿Wide o long?
| Region | ila2010 | ila2011 | ila2012 | ila2013 |
|---|---|---|---|---|
| GBA | 1181.42 | 1200.50 | 1380.58 | 1449.60 |
| Pampeana | 929.61 | 1002.58 | 1223.15 | 1302.65 |
| Cuyo | 769.69 | 780.30 | 850.53 | 927.07 |
| NOA | 692.52 | 705.89 | 875.30 | 901.56 |
| Patagonia | 1505.07 | 1710.60 | 2223.78 | 2490.63 |
| NEA | 613.58 | 652.40 | 841.60 | 967.84 |
Manipular bases: pivot
Para ver un caso, trabajemos con una base ejemplo que tiene para cada región los distintos valores del ingreso laboral en distintos años:
inc_reg <- read.csv("datos/Clase3/inc_region.csv")Visualicemos nuestra base Qué formato tiene ¿Wide o long?
| Region | ila2010 | ila2011 | ila2012 | ila2013 |
|---|---|---|---|---|
| GBA | 1181.42 | 1200.50 | 1380.58 | 1449.60 |
| Pampeana | 929.61 | 1002.58 | 1223.15 | 1302.65 |
| Cuyo | 769.69 | 780.30 | 850.53 | 927.07 |
| NOA | 692.52 | 705.89 | 875.30 | 901.56 |
| Patagonia | 1505.07 | 1710.60 | 2223.78 | 2490.63 |
| NEA | 613.58 | 652.40 | 841.60 | 967.84 |
Wide! Las regiones no se repiten y hay una columna distinta para cada año
Manipular bases: pivot_longer
¿Cómo podemos convertirla a long?
Manipular bases: pivot_longer
¿Cómo podemos convertirla a long?
Hagamos un pivot de nuestra base. Es decir vamos a "alargar" los datos colapsando múltiples columnas del ingreso en una sola y apilando cada uno de sus valores en una misma columna
Manipular bases: pivot_longer
¿Cómo podemos convertirla a long?
Hagamos un pivot de nuestra base. Es decir vamos a "alargar" los datos colapsando múltiples columnas del ingreso en una sola y apilando cada uno de sus valores en una misma columna
Cada una de las columnas que estamos reconviertiendo, son ahora valores por fila de la columna única en la que colapsamos la base
Manipular bases: pivot_longer
¿Cómo podemos convertirla a long?
Hagamos un pivot de nuestra base. Es decir vamos a "alargar" los datos colapsando múltiples columnas del ingreso en una sola y apilando cada uno de sus valores en una misma columna
Cada una de las columnas que estamos reconviertiendo, son ahora valores por fila de la columna única en la que colapsamos la base
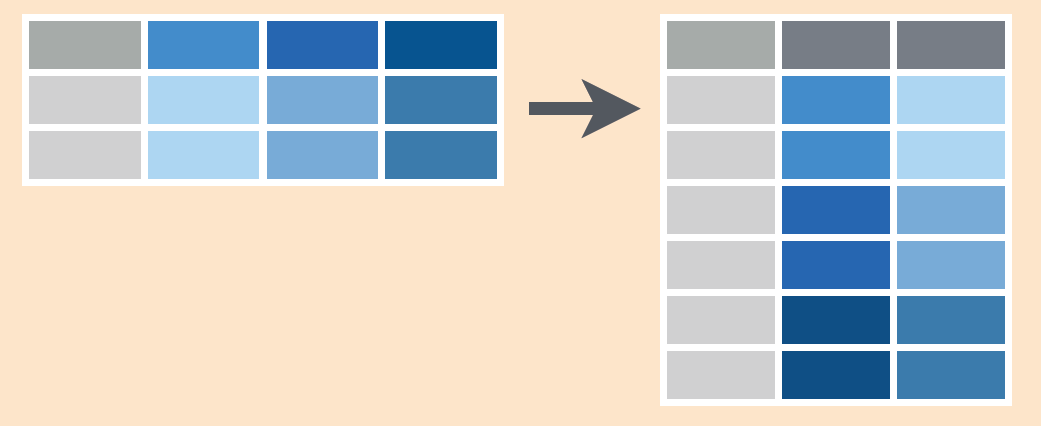
Manipular bases: pivot_longer
La función para realizar esta operación es pivot_longer() y conocer sus argumentos es muy importante:
data
Manipular bases: pivot_longer
La función para realizar esta operación es pivot_longer() y conocer sus argumentos es muy importante:
data:dataframe
Manipular bases: pivot_longer
La función para realizar esta operación es pivot_longer() y conocer sus argumentos es muy importante:
data:dataframecols
Manipular bases: pivot_longer
La función para realizar esta operación es pivot_longer() y conocer sus argumentos es muy importante:
data:dataframecols: columnas que queremos pivotar
Manipular bases: pivot_longer
La función para realizar esta operación es pivot_longer() y conocer sus argumentos es muy importante:
data:dataframecols: columnas que queremos pivotarnames_to = "name"
Manipular bases: pivot_longer
La función para realizar esta operación es pivot_longer() y conocer sus argumentos es muy importante:
data:dataframecols: columnas que queremos pivotarnames_to = "name": nombra la nueva variable que almacena los nombres de las actuales columnas
Manipular bases: pivot_longer
La función para realizar esta operación es pivot_longer() y conocer sus argumentos es muy importante:
data:dataframecols: columnas que queremos pivotarnames_to = "name": nombra la nueva variable que almacena los nombres de las actuales columnasvalues_to = "value"
Manipular bases: pivot_longer
La función para realizar esta operación es pivot_longer() y conocer sus argumentos es muy importante:
data:dataframecols: columnas que queremos pivotarnames_to = "name": nombra la nueva variable que almacena los nombres de las actuales columnasvalues_to = "value": nombra la nueva variable que almacena los valores de cada columna actual
Manipular bases: pivot_longer
La función para realizar esta operación es pivot_longer() y conocer sus argumentos es muy importante:
data:dataframecols: columnas que queremos pivotarnames_to = "name": nombra la nueva variable que almacena los nombres de las actuales columnasvalues_to = "value": nombra la nueva variable que almacena los valores de cada columna actual
Ahora sí, podemos aplicar la función y hacer un reshape de wide a long de la base
inc_reg_long <- inc_reg %>% pivot_longer(cols = c(ila2010, ila2011, ila2012, ila2013), names_to = "anio", values_to = "ila")Manipular bases: pivot_longer
| Region | anio | ila |
|---|---|---|
| GBA | ila2010 | 1181.42 |
| GBA | ila2011 | 1200.50 |
| GBA | ila2012 | 1380.58 |
| GBA | ila2013 | 1449.60 |
| Pampeana | ila2010 | 929.61 |
| Pampeana | ila2011 | 1002.58 |
| Pampeana | ila2012 | 1223.15 |
| Pampeana | ila2013 | 1302.65 |
| Cuyo | ila2010 | 769.69 |
| Cuyo | ila2011 | 780.30 |
| Cuyo | ila2012 | 850.53 |
| Cuyo | ila2013 | 927.07 |
| NOA | ila2010 | 692.52 |
| NOA | ila2011 | 705.89 |
| NOA | ila2012 | 875.30 |
| NOA | ila2013 | 901.56 |
Excelente! Logramos alargar nuestra base
Ahora cada región se repite 4 veces (1 por año)
--
Tenemos una columna que condensa lo que antes eran nombres de columnas separadas
--
Tenemos una columna que condensa los valores de las columnas anteriores
Manipular bases: pivot_longer
Veamos un argumento adicional que suele ser muy útil. De la tabla de datos pueden notar que la columna año contiene el string "ila" seguido por el año en números
Manipular bases: pivot_longer
Veamos un argumento adicional que suele ser muy útil. De la tabla de datos pueden notar que la columna año contiene el string "ila" seguido por el año en númerosPara tener una base limpia no quisieramos repetir ese string, sino solo quedarnos con una columna numérica de años.
Manipular bases: pivot_longer
Veamos un argumento adicional que suele ser muy útil. De la tabla de datos pueden notar que la columna año contiene el string "ila" seguido por el año en númerosPara tener una base limpia no quisieramos repetir ese string, sino solo quedarnos con una columna numérica de años.
La función posee un argumento opcional names_prefix= que justamente hace este trabajo, tomando el patron indicado y quitandolo de los valores de la nueva columna
Manipular bases: pivot_longer
Veamos un argumento adicional que suele ser muy útil. De la tabla de datos pueden notar que la columna año contiene el string "ila" seguido por el año en númerosPara tener una base limpia no quisieramos repetir ese string, sino solo quedarnos con una columna numérica de años.
La función posee un argumento opcional names_prefix= que justamente hace este trabajo, tomando el patron indicado y quitandolo de los valores de la nueva columna
inc_reg_long_v2 <- inc_reg %>% pivot_longer(cols = starts_with("ila"), #check this names_to = "anio", names_prefix = "ila", values_to = "ila")Manipular bases: pivot_longer
Veamos un argumento adicional que suele ser muy útil. De la tabla de datos pueden notar que la columna año contiene el string "ila" seguido por el año en númerosPara tener una base limpia no quisieramos repetir ese string, sino solo quedarnos con una columna numérica de años.
La función posee un argumento opcional names_prefix= que justamente hace este trabajo, tomando el patron indicado y quitandolo de los valores de la nueva columna
inc_reg_long_v2 <- inc_reg %>% pivot_longer(cols = starts_with("ila"), #check this names_to = "anio", names_prefix = "ila", values_to = "ila")Ahora comparemos resultados y veamos como mejora nuestra tabla
Manipular bases: pivot_longer
Antes:
| Region | anio | ila |
|---|---|---|
| GBA | ila2010 | 1181.42 |
| GBA | ila2011 | 1200.50 |
| GBA | ila2012 | 1380.58 |
| GBA | ila2013 | 1449.60 |
| Pampeana | ila2010 | 929.61 |
| Pampeana | ila2011 | 1002.58 |
| Pampeana | ila2012 | 1223.15 |
| Pampeana | ila2013 | 1302.65 |
| Cuyo | ila2010 | 769.69 |
| Cuyo | ila2011 | 780.30 |
| Cuyo | ila2012 | 850.53 |
| Cuyo | ila2013 | 927.07 |
| NOA | ila2010 | 692.52 |
| NOA | ila2011 | 705.89 |
| NOA | ila2012 | 875.30 |
Después:
| Region | anio | ila |
|---|---|---|
| GBA | 2010 | 1181.42 |
| GBA | 2011 | 1200.50 |
| GBA | 2012 | 1380.58 |
| GBA | 2013 | 1449.60 |
| Pampeana | 2010 | 929.61 |
| Pampeana | 2011 | 1002.58 |
| Pampeana | 2012 | 1223.15 |
| Pampeana | 2013 | 1302.65 |
| Cuyo | 2010 | 769.69 |
| Cuyo | 2011 | 780.30 |
| Cuyo | 2012 | 850.53 |
| Cuyo | 2013 | 927.07 |
| NOA | 2010 | 692.52 |
| NOA | 2011 | 705.89 |
| NOA | 2012 | 875.30 |
Manipular bases: pivot_wider
Bien, ahora que tenemos nuestra base en formato long, aprendamos como pasar a wide. Queremos ahora "ampliar" la base de datos expandiendo una columna en varias.
Para un mismo grupo, cada valor distinto que tomaba la columna que estamos reconviertiendo, es ahora una columna de la base resultante
Manipular bases: pivot_wider
Bien, ahora que tenemos nuestra base en formato long, aprendamos como pasar a wide. Queremos ahora "ampliar" la base de datos expandiendo una columna en varias.
Para un mismo grupo, cada valor distinto que tomaba la columna que estamos reconviertiendo, es ahora una columna de la base resultante

Manipular bases: pivot_wider
La función para realizar esta operación es pivot_wider y sus argumentos son distintos a la función anterior
Manipular bases: pivot_wider
La función para realizar esta operación es pivot_wider y sus argumentos son distintos a la función anterior
data
Manipular bases: pivot_wider
La función para realizar esta operación es pivot_wider y sus argumentos son distintos a la función anterior
data: indica el dataframe
Manipular bases: pivot_wider
La función para realizar esta operación es pivot_wider y sus argumentos son distintos a la función anterior
data: indica el dataframenames_from = "name"
Manipular bases: pivot_wider
La función para realizar esta operación es pivot_wider y sus argumentos son distintos a la función anterior
data: indica el dataframenames_from = "name": qué columna buscamos convertir en múltiples columnas
Manipular bases: pivot_wider
La función para realizar esta operación es pivot_wider y sus argumentos son distintos a la función anterior
data: indica el dataframenames_from = "name": qué columna buscamos convertir en múltiples columnasvalues_from = "value"
Manipular bases: pivot_wider
La función para realizar esta operación es pivot_wider y sus argumentos son distintos a la función anterior
data: indica el dataframenames_from = "name": qué columna buscamos convertir en múltiples columnasvalues_from = "value": qué columna almacena los valores de las nuevas columnas
Manipular bases: pivot_wider
La función para realizar esta operación es pivot_wider y sus argumentos son distintos a la función anterior
data: indica el dataframenames_from = "name": qué columna buscamos convertir en múltiples columnasvalues_from = "value": qué columna almacena los valores de las nuevas columnas
Apliquemos la función para pasar de long a wide
inc_reg_wide <- inc_reg_long %>% pivot_wider(names_from = anio, values_from = ila )Manipular bases: pivot_wider
Veamos cómo resultó
Manipular bases: pivot_wider
Veamos cómo resultó
| Region | ila2010 | ila2011 | ila2012 | ila2013 |
|---|---|---|---|---|
| GBA | 1181.42 | 1200.50 | 1380.58 | 1449.60 |
| Pampeana | 929.61 | 1002.58 | 1223.15 | 1302.65 |
| Cuyo | 769.69 | 780.30 | 850.53 | 927.07 |
| NOA | 692.52 | 705.89 | 875.30 | 901.56 |
| Patagonia | 1505.07 | 1710.60 | 2223.78 | 2490.63 |
| NEA | 613.58 | 652.40 | 841.60 | 967.84 |
Bien, volvimos a nuestro formato wide original:
- Las regiones no se repiten
Manipular bases: pivot_wider
Veamos cómo resultó
| Region | ila2010 | ila2011 | ila2012 | ila2013 |
|---|---|---|---|---|
| GBA | 1181.42 | 1200.50 | 1380.58 | 1449.60 |
| Pampeana | 929.61 | 1002.58 | 1223.15 | 1302.65 |
| Cuyo | 769.69 | 780.30 | 850.53 | 927.07 |
| NOA | 692.52 | 705.89 | 875.30 | 901.56 |
| Patagonia | 1505.07 | 1710.60 | 2223.78 | 2490.63 |
| NEA | 613.58 | 652.40 | 841.60 | 967.84 |
Bien, volvimos a nuestro formato wide original:
Las regiones no se repiten
Y para cada año tenemos una columna
Manipular bases: pivot
Hasta ahora vimos un caso relativamente simple, siempre condensamos muchas columnas en una sola
Manipular bases: pivot
Hasta ahora vimos un caso relativamente simple, siempre condensamos muchas columnas en una sola . Suele darse casos mas desafiantes, donde la estructura de los datos requiere realizar un reshape hacia multiples columnas
Manipular bases: pivot
Hasta ahora vimos un caso relativamente simple, siempre condensamos muchas columnas en una sola . Suele darse casos mas desafiantes, donde la estructura de los datos requiere realizar un reshape hacia multiples columnas
Para ver un ejemplo, carguemos también una base de ingreso a nivel de regiones, pero ahora tenemos mas columnas:
inc_reg_ext <- read.csv("datos/Clase3/inc_region_ext.csv", sep=";")| Region | ila_2010_ars | ila_2011_ars | ila_2012_ars | ila_2013_ars | ila_2010_usd | ila_2011_usd | ila_2012_usd | ila_2013_usd | inola_2010_ars | inola_2011_ars | inola_2012_ars | inola_2013_ars | inola_2010_usd | inola_2011_usd | inola_2012_usd | inola_2013_usd |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| GBA | 118142 | 12005 | 138058 | 14496 | 303 | 31 | 354 | 37 | 23628 | 2401 | 27612 | 2899 | 61 | 6 | 71 | 7 |
| Pampeana | 92961 | 100258 | 122315 | 130265 | 238 | 257 | 314 | 334 | 18592 | 20052 | 24463 | 26053 | 48 | 51 | 63 | 67 |
| Cuyo | 76969 | 7803 | 85053 | 92707 | 197 | 20 | 218 | 238 | 15394 | 1561 | 17011 | 18541 | 39 | 4 | 44 | 48 |
| NOA | 69252 | 70589 | 8753 | 90156 | 178 | 181 | 22 | 231 | 13850 | 14118 | 1751 | 18031 | 36 | 36 | 4 | 46 |
| Patagonia | 150507 | 17106 | 222378 | 249063 | 386 | 44 | 570 | 639 | 30101 | 3421 | 44476 | 49813 | 77 | 9 | 114 | 128 |
| NEA | 61358 | 6524 | 8416 | 96784 | 157 | 17 | 22 | 248 | 12272 | 1305 | 1683 | 19357 | 31 | 3 | 4 | 50 |
Manipular bases: pivot
Ahora ya no solo tenemos el ingreso laboral para distintos años, sino que en este nuevo dataframe los nombres de las columnas indican además del año otras distintas opciones para el ingreso:
- ingreso laboral e ingreso no laboral
- expresado en pesos o en dolares
Manipular bases: pivot
Ahora ya no solo tenemos el ingreso laboral para distintos años, sino que en este nuevo dataframe los nombres de las columnas indican además del año otras distintas opciones para el ingreso:
- ingreso laboral e ingreso no laboral
- expresado en pesos o en dolares
Tenemos 4 años, 2 tipos de monedas y 2 tipos de ingreso, en total son 16 columnas
Manipular bases: pivot
Ahora ya no solo tenemos el ingreso laboral para distintos años, sino que en este nuevo dataframe los nombres de las columnas indican además del año otras distintas opciones para el ingreso:
- ingreso laboral e ingreso no laboral
- expresado en pesos o en dolares
Tenemos 4 años, 2 tipos de monedas y 2 tipos de ingreso, en total son 16 columnas
Que podemos re-organizar en 3 columnas diferentes: "ingreso", "años" y "moneda"
Manipular bases: pivot
Ahora ya no solo tenemos el ingreso laboral para distintos años, sino que en este nuevo dataframe los nombres de las columnas indican además del año otras distintas opciones para el ingreso:
- ingreso laboral e ingreso no laboral
- expresado en pesos o en dolares
Tenemos 4 años, 2 tipos de monedas y 2 tipos de ingreso, en total son 16 columnas
Que podemos re-organizar en 3 columnas diferentes: "ingreso", "años" y "moneda"
¿Cómo lograr identifcar esta separación?. Cada una de las opciones está indicada luego de un "_"
Manipular bases: pivot
Ahora ya no solo tenemos el ingreso laboral para distintos años, sino que en este nuevo dataframe los nombres de las columnas indican además del año otras distintas opciones para el ingreso:
- ingreso laboral e ingreso no laboral
- expresado en pesos o en dolares
Tenemos 4 años, 2 tipos de monedas y 2 tipos de ingreso, en total son 16 columnas
Que podemos re-organizar en 3 columnas diferentes: "ingreso", "años" y "moneda"
¿Cómo lograr identifcar esta separación?. Cada una de las opciones está indicada luego de un "_"
Este patrón nos permite seprar las columnas -- Veamos como escribir el código:
Manipular bases: pivot
Ahora queremos pivotear todas las columnas y mantener la de región, esto mismo es lo que especificamos en el código.
Manipular bases: pivot
Ahora queremos pivotear todas las columnas y mantener la de región, esto mismo es lo que especificamos en el código.
El argumento clave es names_sep="_"
inc_reg_long <- inc_reg_ext %>% pivot_longer( cols = !(Region), names_to = c("ingreso", "año", "moneda"), names_sep = "_", values_to = "monto" )Manipular bases: pivot
Veamos nuestro resultado
| Region | ingreso | año | moneda | monto |
|---|---|---|---|---|
| GBA | ila | 2010 | ars | 118142 |
| GBA | ila | 2011 | ars | 12005 |
| GBA | ila | 2012 | ars | 138058 |
| GBA | ila | 2013 | ars | 14496 |
| GBA | ila | 2010 | usd | 303 |
| GBA | ila | 2011 | usd | 31 |
| GBA | ila | 2012 | usd | 354 |
| GBA | ila | 2013 | usd | 37 |
| GBA | inola | 2010 | ars | 23628 |
| GBA | inola | 2011 | ars | 2401 |
| GBA | inola | 2012 | ars | 27612 |
| GBA | inola | 2013 | ars | 2899 |
| GBA | inola | 2010 | usd | 61 |
| GBA | inola | 2011 | usd | 6 |
| GBA | inola | 2012 | usd | 71 |
| GBA | inola | 2013 | usd | 7 |
| Pampeana | ila | 2010 | ars | 92961 |
| Pampeana | ila | 2011 | ars | 100258 |
| Pampeana | ila | 2012 | ars | 122315 |
| Pampeana | ila | 2013 | ars | 130265 |
| Pampeana | ila | 2010 | usd | 238 |
| Pampeana | ila | 2011 | usd | 257 |
| Pampeana | ila | 2012 | usd | 314 |
| Pampeana | ila | 2013 | usd | 334 |
| Pampeana | inola | 2010 | ars | 18592 |
| Pampeana | inola | 2011 | ars | 20052 |
| Pampeana | inola | 2012 | ars | 24463 |
| Pampeana | inola | 2013 | ars | 26053 |
| Pampeana | inola | 2010 | usd | 48 |
| Pampeana | inola | 2011 | usd | 51 |
| Pampeana | inola | 2012 | usd | 63 |
| Pampeana | inola | 2013 | usd | 67 |
| Cuyo | ila | 2010 | ars | 76969 |
| Cuyo | ila | 2011 | ars | 7803 |
| Cuyo | ila | 2012 | ars | 85053 |
| Cuyo | ila | 2013 | ars | 92707 |
| Cuyo | ila | 2010 | usd | 197 |
| Cuyo | ila | 2011 | usd | 20 |
| Cuyo | ila | 2012 | usd | 218 |
| Cuyo | ila | 2013 | usd | 238 |
| Cuyo | inola | 2010 | ars | 15394 |
| Cuyo | inola | 2011 | ars | 1561 |
| Cuyo | inola | 2012 | ars | 17011 |
| Cuyo | inola | 2013 | ars | 18541 |
| Cuyo | inola | 2010 | usd | 39 |
| Cuyo | inola | 2011 | usd | 4 |
| Cuyo | inola | 2012 | usd | 44 |
| Cuyo | inola | 2013 | usd | 48 |
| NOA | ila | 2010 | ars | 69252 |
| NOA | ila | 2011 | ars | 70589 |
| NOA | ila | 2012 | ars | 8753 |
| NOA | ila | 2013 | ars | 90156 |
| NOA | ila | 2010 | usd | 178 |
| NOA | ila | 2011 | usd | 181 |
| NOA | ila | 2012 | usd | 22 |
| NOA | ila | 2013 | usd | 231 |
| NOA | inola | 2010 | ars | 13850 |
| NOA | inola | 2011 | ars | 14118 |
| NOA | inola | 2012 | ars | 1751 |
| NOA | inola | 2013 | ars | 18031 |
| NOA | inola | 2010 | usd | 36 |
| NOA | inola | 2011 | usd | 36 |
| NOA | inola | 2012 | usd | 4 |
| NOA | inola | 2013 | usd | 46 |
| Patagonia | ila | 2010 | ars | 150507 |
| Patagonia | ila | 2011 | ars | 17106 |
| Patagonia | ila | 2012 | ars | 222378 |
| Patagonia | ila | 2013 | ars | 249063 |
| Patagonia | ila | 2010 | usd | 386 |
| Patagonia | ila | 2011 | usd | 44 |
| Patagonia | ila | 2012 | usd | 570 |
| Patagonia | ila | 2013 | usd | 639 |
| Patagonia | inola | 2010 | ars | 30101 |
| Patagonia | inola | 2011 | ars | 3421 |
| Patagonia | inola | 2012 | ars | 44476 |
| Patagonia | inola | 2013 | ars | 49813 |
| Patagonia | inola | 2010 | usd | 77 |
| Patagonia | inola | 2011 | usd | 9 |
| Patagonia | inola | 2012 | usd | 114 |
| Patagonia | inola | 2013 | usd | 128 |
| NEA | ila | 2010 | ars | 61358 |
| NEA | ila | 2011 | ars | 6524 |
| NEA | ila | 2012 | ars | 8416 |
| NEA | ila | 2013 | ars | 96784 |
| NEA | ila | 2010 | usd | 157 |
| NEA | ila | 2011 | usd | 17 |
| NEA | ila | 2012 | usd | 22 |
| NEA | ila | 2013 | usd | 248 |
| NEA | inola | 2010 | ars | 12272 |
| NEA | inola | 2011 | ars | 1305 |
| NEA | inola | 2012 | ars | 1683 |
| NEA | inola | 2013 | ars | 19357 |
| NEA | inola | 2010 | usd | 31 |
| NEA | inola | 2011 | usd | 3 |
| NEA | inola | 2012 | usd | 4 |
| NEA | inola | 2013 | usd | 50 |
Gestionar bases:
Para finalizar veamos como importar y exportar multiples tipos de bases de datos, y cómo gestionar los path y carpetas para trabajar ordenadamente
El primer paso para gestionar bases de datos es poder ordenarnos:
Gestionar bases:
Para finalizar veamos como importar y exportar multiples tipos de bases de datos, y cómo gestionar los path y carpetas para trabajar ordenadamente
El primer paso para gestionar bases de datos es poder ordenarnos:
Para eso nos resulta de ayuda gestionar los directorios desde los cuales vamos a trabajar.
Gestionar bases:
Para finalizar veamos como importar y exportar multiples tipos de bases de datos, y cómo gestionar los path y carpetas para trabajar ordenadamente
El primer paso para gestionar bases de datos es poder ordenarnos:
Para eso nos resulta de ayuda gestionar los directorios desde los cuales vamos a trabajar.
Gestionar bases: directorio
Una primera opción es fijar los directorios con las funciones
-setwd(): permite fijar el directorio de trabajo
-getwd(): devuelve el directorio actual de trabajo
#defino pathsetwd("C:/Users/HP/Dropbox/Educ Mobility - Censos IPUMS/")#return actual pathgetwd()Gestionar bases: directorio
Una primera opción es fijar los directorios con las funciones
-setwd(): permite fijar el directorio de trabajo
-getwd(): devuelve el directorio actual de trabajo
#defino pathsetwd("C:/Users/HP/Dropbox/Educ Mobility - Censos IPUMS/")#set folders pathdata_dir <- paste(getwd(), "Data/") script_dir <- paste(getwd(), "Codes/R-scripts/auxiliar codes/")Gestionar bases: directorio
Un tip que utilizo mucho es definir una simbolo que cumpla la función de paste, de concatenar strings pero de manera mas directa
Gestionar bases: directorio
Un tip que utilizo mucho es definir una simbolo que cumpla la función de paste, de concatenar strings pero de manera mas directa
Como si fuera un "pipe para strings"
"%+%" <- function(x,y) paste(x,y,sep = "") #defino pathsetwd("C:/Users/HP/Dropbox/Educ Mobility - Censos IPUMS/")#set folders pathdata_dir <- getwd() %+% "Data/") script_dir <- getwd() %+% "Codes/R-scripts/auxiliar codes/")Gestionar bases: directorio
Otra opción es definir el path en un objeto y luego llamarlo cuando vayamos a acceder a un archivo o para setear subcarpetas
Gestionar bases: directorio
Otra opción es definir el path en un objeto y luego llamarlo cuando vayamos a acceder a un archivo o para setear subcarpetas
Definamos el objeto root como path general y luego las subcarpetas donde queremos guardar las bases y exportar los resultados
"%+%" <- function(x,y) paste(x,y,sep = "") #defino pathroot <- "C:/Users/HP/Dropbox/Educ Mobility - Censos IPUMS/"#set folders pathdata_dir <- root %+% "Data/" script_dir <- root %+% "Codes/R-scripts/auxiliar codes/"Noten que, a diferencia de usar setwd(), en esta alternativa cada vez que quieran moverse en la carpeta deben invocar a root.
Gestionar bases: directorio
Otra opción es definir el path en un objeto y luego llamarlo cuando vayamos a acceder a un archivo o para setear subcarpetas
Definamos el objeto root como path general y luego las subcarpetas donde queremos guardar las bases y exportar los resultados
"%+%" <- function(x,y) paste(x,y,sep = "") #defino pathroot <- "C:/Users/HP/Dropbox/Educ Mobility - Censos IPUMS/"#set folders pathdata_dir <- root %+% "Data/" script_dir <- root %+% "Codes/R-scripts/auxiliar codes/"Noten que, a diferencia de usar setwd(), en esta alternativa cada vez que quieran moverse en la carpeta deben invocar a root.
Ese directorio queda en un objeto y no como directorio por defecto, a menos que lo fijen con setwd()
Gestionar bases: importar
En la segunda clase vimos dos funciones funciones de R base para importar base de datos
read.csv()read.xls()
Gestionar bases: importar
En la segunda clase vimos dos funciones funciones de R base para importar base de datos
read.csv()read.xls()
Para cargar archivos en extensión csv, las funciones básicas funcionan bien, y estos archivos son bastante estandard
Gestionar bases: importar
En la segunda clase vimos dos funciones funciones de R base para importar base de datos
read.csv()read.xls()
Para cargar archivos en extensión csv, las funciones básicas funcionan bien, y estos archivos son bastante estandard
Para archivos de Excel es recomendable sumar la librería readxl, que contempla distintos formatos
library(readxl)eph_ind_old <- read_xls(data_dir %+% "/Clase3/EPH_3T_22/usu_individual_T322.xls")eph_ind_new <- read_xlsx(data_dir %+% "/Clase3/EPH_3T_22/usu_individual_T322.xlsx")Gestionar bases: importar
Para otros formatos externos, precisamos la libreria haven, que incorpora funciones read para extensiones no nativas
library(haven)eph_ind_stat <- read_dta(data_dir %+% "/Clase3/EPH_3T_22/usu_individual_T322.dta")eph_ind_sas <- read_sas(data_dir %+% "/Clase3/EPH_3T_22/usu_individual_T322.sas")eph_ind_spss <- read_spss(data_dir %+% "/Clase3/EPH_3T_22/usu_individual_T322.spss")Gestionar bases: importar
Pero no está demás aclarar que también existe el formato nativo de R, es decir, podemos cargar y exportar bases en el formato "original" de R
Gestionar bases: importar
Pero no está demás aclarar que también existe el formato nativo de R, es decir, podemos cargar y exportar bases en el formato "original" de REstos formatos son dos: .Rdata y RDS
eph_ind_rdata <- load(data_dir %+% "/Clase3/eph_R.RData")eph_ind_rds <- readRDS(data_dir %+% "/Clase3/eph_R.RDS")Gestionar bases: exportar
Para exportar en formato R, también es muy simple, con las funciones save() y saveRDS()
save(df, file = ouput_dir %+% "df.RData")saveRDS(df, file = ouput_dir %+% "df.RDS")Gestionar bases: exportar
Para exportar en formato R, también es muy simple, con las funciones save() y saveRDS()
save(df, file = ouput_dir %+% "df.RData")saveRDS(df, file = ouput_dir %+% "df.RDS")Para exportar en un lenguaje no nativo también usamos la librería haven, con las funciónes del tipo write_
library(haven)write_dta(df, output_dir %+% "/Clase3/EPH_3T_22/usu_individual_T322.dta")write_dta_sas(df, output_dir %+% "/Clase3/EPH_3T_22/usu_individual_T322.sas")write_dta_spss(df, output_dir %+% "/Clase3/EPH_3T_22/usu_individual_T322.spss")Gestionar bases: exportar
Lo mismo aplica para exportar un archivo de texto
write.csv(df, output_dir %+% "/Clase3/EPH_3T_22/usu_individual_T322.csv")Gestionar bases: exportar
Lo mismo aplica para exportar un archivo de texto
write.csv(df, output_dir %+% "/Clase3/EPH_3T_22/usu_individual_T322.csv")Para exportar un archivo en excel, así como para importar usábamos readxl para exportar existe la librería writexl
write_xlsx(df, output_dir %+% "/Clase3/EPH_3T_22/usu_individual_T322.xlsx")Gestionar bases: exportar
Un uso muy útil de esta función es cuando queremos exportar múltiples dataframes en distintas hojas
Para ello, definimos una lista, donde especificamos el nombre de la hoja de excel y el dataframe que exportamos allí
write_xlsx(list( "Estimación A" = df_1, "Estimación B" = df_2, "Estimación C" = df_3 ) output_dir %+% "/Clase3/EPH_3T_22/usu_individual_T322.xlsx")Gestionar bases: exportar
Dado que existen muchas funciones distintas para importar y cargar, siempre es bueno estar atentos a que la función que usamos proviene de la libería que queremos usar, y no de otra que tiene una función nombre similar
Gestionar bases: exportar
Dado que existen muchas funciones distintas para importar y cargar, siempre es bueno estar atentos a que la función que usamos proviene de la libería que queremos usar, y no de otra que tiene una función nombre similar
Sino directamente podemos llamar a las funciones de esa librería
eph_ind_new <- readxl::read_xlsx(data_dir %+% "/Clase3/EPH_3T_22/usu_individual_T322.xlsx")writexl::write_xlsx(df, output_dir %+% "/Clase3/EPH_3T_22/usu_individual_T322.xlsx")Gestionar bases: Workflow y repaso
Ahora que sabemos declarar directorios, manejar subcarpetas, importar y exportar en múltiples formatos, hagamos un repaso de cómo sería un workflow completo en un código de R
Gestionar bases: Workflow y repaso
Ahora que sabemos declarar directorios, manejar subcarpetas, importar y exportar en múltiples formatos, hagamos un repaso de cómo sería un workflow completo en un código de R
Para ello abrimos el archivo scipt_modelo