Seminario de Instrumentos Computacionales
R - Clase 2
Cristian Bonavida
Repaso Clase 1
En la clase anterior vimos
- Qué era R y cuáles eran sus ventajas comparativas
Repaso Clase 1
En la clase anterior vimos
- Qué era R y cuáles eran sus ventajas comparativas
- Aprendimos los fundamentos de R, es decir el ABC de este lenguaje
- valores
- vectores
- funciones
- dataframes
Repaso Clase 1
En la clase anterior vimos
- Qué era R y cuáles eran sus ventajas comparativas
- Aprendimos los fundamentos de R, es decir el ABC de este lenguaje
- valores
- vectores
- funciones
- dataframes
- Vimos qué tipos de datos existen y cómo reconocerlos
Repaso Clase 1
En la clase anterior vimos
- Qué era R y cuáles eran sus ventajas comparativas
- Aprendimos los fundamentos de R, es decir el ABC de este lenguaje
- valores
- vectores
- funciones
- dataframes
- Vimos qué tipos de datos existen y cómo reconocerlos
- Definimos un dataframe y vimos cómo navegar por él, llamando filas y columnas, filtrando observaciones y usando el operador
$
Data frames y bases de datos
En la gran mayoría de las veces, no definimos un dataframe a mano, sino que este es una base de datos que importamos de un archivo, tal como ocurre con las encuestas de hogares.
Los misma sintaxis que vimos antes nos servirán para manipular bases de datos mas grandes
El primer paso es saber importarlas
Importar bases
R es muy eficiente importando bases de datos en múltiples formatos, incluso desde otros lenguajes
Veamos el caso de la EPH. Cuando la descargamos de INDEC podemos bajar el archivo en formato txt y en formato xls. Para leer respectivas extensiones podemos usar las funciones de R base:
read.csv()read.xls()
Importar bases
R es muy eficiente importando bases de datos en múltiples formatos, incluso desde otros lenguajes
Veamos el caso de la EPH. Cuando la descargamos de INDEC podemos bajar el archivo en formato txt y en formato xls. Para leer respectivas extensiones podemos usar las funciones de R base:
read.csv()read.xls()
read.csv("datos/Clase2/EPH_3T_22/usu_individual_T322.txt", sep=";", dec=",")sep=con este argumento indicamos cuál es el patrón que separa cada columna, cada vez que R encuentre en el archivo de texto ";" sabrá que allí comienza una nueva columnadec=le indica que los decimales en el archivo están indicados por la coma y no por el punto. Si no indicamos esto R entiende la columna como texto y al pasarla a numérica genera casosNAs
Importar bases
Notar que solo la importamos, por lo tanto el output del comando (omitido aquí) será un print de las primeras filas.
Recuerden que en R podemos tener múltiples dataframes cargados en el entorno de trabajo, por eso, lo que debemos hacer es asignar esta base de datos a un objeto, es decir almacenarla con un nombre
eph_ind <- read.csv("datos/Clase2/EPH_3T_22/usu_individual_T322.txt", sep=";", dec=",")Ahora R no imprime nada, si la carga fue exitosa no obtendremos ningún output al correr el código
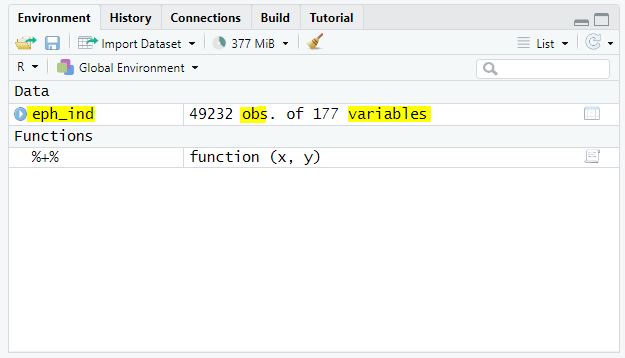
Importar bases
Pero en nuestro entorno de trabajo debemos ver ahora la base cargada, indicando cantidad filas (obs) y columnas (variables)
Explorar la base de datos
Cuántas filas y cuantas columnas tiene nuestra base
dim(eph_ind)## [1] 49232 177Cuáles son los nombres de esas columnas
names(eph_ind)## [1] "CODUSU" "ANO4" "TRIMESTRE" "NRO_HOGAR" "COMPONENTE"## [6] "H15" "REGION" "MAS_500" "AGLOMERADO" "PONDERA" ## [11] "CH03" "CH04" "CH05" "CH06" "CH07" ## [16] "CH08" "CH09" "CH10" "CH11" "CH12" ## [21] "CH13" "CH14" "CH15" "CH15_COD" "CH16" ## [26] "CH16_COD" "NIVEL_ED" "ESTADO" "CAT_OCUP" "CAT_INAC" ## [31] "IMPUTA" "PP02C1" "PP02C2" "PP02C3" "PP02C4" ## [36] "PP02C5" "PP02C6" "PP02C7" "PP02C8" "PP02E" ## [41] "PP02H" "PP02I" "PP03C" "PP03D" "PP3E_TOT" ## [46] "PP3F_TOT" "PP03G" "PP03H" "PP03I" "PP03J" ## [51] "INTENSI" "PP04A" "PP04B_COD" "PP04B1" "PP04B2" ## [56] "PP04B3_MES" "PP04B3_ANO" "PP04B3_DIA" "PP04C" "PP04C99" ## [61] "PP04D_COD" "PP04G" "PP05B2_MES" "PP05B2_ANO" "PP05B2_DIA"## [66] "PP05C_1" "PP05C_2" "PP05C_3" "PP05E" "PP05F" ## [71] "PP05H" "PP06A" "PP06C" "PP06D" "PP06E" ## [76] "PP06H" "PP07A" "PP07C" "PP07D" "PP07E" ## [81] "PP07F1" "PP07F2" "PP07F3" "PP07F4" "PP07F5" ## [86] "PP07G1" "PP07G2" "PP07G3" "PP07G4" "PP07G_59" ## [91] "PP07H" "PP07I" "PP07J" "PP07K" "PP08D1" ## [96] "PP08D4" "PP08F1" "PP08F2" "PP08J1" "PP08J2" ## [101] "PP08J3" "PP09A" "PP09A_ESP" "PP09B" "PP09C" ## [106] "PP09C_ESP" "PP10A" "PP10C" "PP10D" "PP10E" ## [111] "PP11A" "PP11B_COD" "PP11B1" "PP11B2_MES" "PP11B2_ANO"## [116] "PP11B2_DIA" "PP11C" "PP11C99" "PP11D_COD" "PP11G_ANO" ## [121] "PP11G_MES" "PP11G_DIA" "PP11L" "PP11L1" "PP11M" ## [126] "PP11N" "PP11O" "PP11P" "PP11Q" "PP11R" ## [131] "PP11S" "PP11T" "P21" "DECOCUR" "IDECOCUR" ## [136] "RDECOCUR" "GDECOCUR" "PDECOCUR" "ADECOCUR" "PONDIIO" ## [141] "TOT_P12" "P47T" "DECINDR" "IDECINDR" "RDECINDR" ## [146] "GDECINDR" "PDECINDR" "ADECINDR" "PONDII" "V2_M" ## [151] "V3_M" "V4_M" "V5_M" "V8_M" "V9_M" ## [156] "V10_M" "V11_M" "V12_M" "V18_M" "V19_AM" ## [161] "V21_M" "T_VI" "ITF" "DECIFR" "IDECIFR" ## [166] "RDECIFR" "GDECIFR" "PDECIFR" "ADECIFR" "IPCF" ## [171] "DECCFR" "IDECCFR" "RDECCFR" "GDECCFR" "PDECCFR" ## [176] "ADECCFR" "PONDIH"Explorar la base de datos
Quiero chequear que todo esté ok, para eso puedo imprimir las primeras n filas (n=10 en este caso)
head(eph_ind, 10)## CODUSU ANO4 TRIMESTRE NRO_HOGAR COMPONENTE H15 REGION## 1 TQRMNORVQHJNLTCDEGGFB00788741 2022 3 1 1 1 42## 2 TQRMNOQSWHKNKRCDEGJBF00789123 2022 3 1 1 1 43## 3 TQRMNOQSWHKNKRCDEGJBF00789123 2022 3 1 2 1 43## 4 TQRMNOTSQHLLMLCDEIIAD00703213 2022 3 1 1 1 1## 5 TQRMNOTSQHLLMLCDEIIAD00703213 2022 3 1 2 1 1## 6 TQRMNOSWVHKMKOCDEHPJB00790947 2022 3 1 1 1 40## 7 TQRMNOSWVHKMKOCDEHPJB00790947 2022 3 1 2 1 40## 8 TQRMNOSWVHKMKOCDEHPJB00790947 2022 3 1 3 1 40## 9 TQRMNOSWVHKMKOCDEHPJB00790947 2022 3 1 4 0 40## 10 TQRMNOSWVHKMKOCDEHPJB00790947 2022 3 1 5 1 40## MAS_500 AGLOMERADO PONDERA CH03 CH04 CH05 CH06 CH07 CH08 CH09 CH10## 1 S 10 537 1 1 10/04/1961 61 5 3 1 2## 2 S 13 543 1 1 06/10/1953 68 2 1 1 2## 3 S 13 543 2 2 28/08/1955 66 2 1 1 2## 4 S 32 894 1 1 10/03/1957 65 2 12 1 2## 5 S 32 894 2 2 07/01/1958 64 2 12 1 2## 6 S 29 345 1 2 18/09/1977 44 5 4 1 2## 7 S 29 345 3 1 13/05/2003 19 1 4 1 2## 8 S 29 345 3 1 30/03/2010 12 5 4 1 1## 9 S 29 345 3 1 06/03/2015 7 5 3 2 1## 10 S 29 345 4 2 30/04/2005 17 1 4 1 2## CH11 CH12 CH13 CH14 CH15 CH15_COD CH16 CH16_COD NIVEL_ED ESTADO CAT_OCUP## 1 0 4 2 2 1 NA 1 NA 3 1 2## 2 0 4 1 NA 1 NA 1 NA 4 3 0## 3 0 7 1 NA 1 NA 1 NA 6 3 0## 4 0 8 1 NA 1 NA 1 NA 6 3 0## 5 0 6 1 NA 1 NA 1 NA 6 3 0## 6 0 2 2 4 1 NA 1 NA 1 3 0## 7 0 4 2 1 1 NA 1 NA 3 1 3## 8 1 4 2 0 1 NA 1 NA 3 3 0## 9 1 2 2 1 1 NA 1 NA 1 4 0## 10 0 4 2 1 1 NA 1 NA 3 1 3## CAT_INAC IMPUTA PP02C1 PP02C2 PP02C3 PP02C4 PP02C5 PP02C6 PP02C7 PP02C8## 1 0 NA 0 0 0 0 0 0 0 0## 2 1 NA 0 0 0 0 0 0 0 0## 3 1 NA 0 0 0 0 0 0 0 0## 4 1 NA 0 0 0 0 0 0 0 0## 5 1 NA 0 0 0 0 0 0 0 0## 6 4 NA 0 0 0 0 0 0 0 0## 7 0 NA 0 0 0 0 0 0 0 0## 8 3 NA 0 0 0 0 0 0 0 0## 9 1 NA 0 0 0 0 0 0 0 0## 10 0 NA 0 0 0 0 0 0 0 0## PP02E PP02H PP02I PP03C PP03D PP3E_TOT PP3F_TOT PP03G PP03H PP03I PP03J## 1 0 0 0 1 0 32 0 1 1 1 1## 2 0 2 2 NA NA NA NA NA NA NA NA## 3 0 2 2 NA NA NA NA NA NA NA NA## 4 0 2 2 NA NA NA NA NA NA NA NA## 5 0 2 2 NA NA NA NA NA NA NA NA## 6 0 2 2 NA NA NA NA NA NA NA NA## 7 0 0 0 1 0 8 0 1 1 1 2## 8 0 2 2 NA NA NA NA NA NA NA NA## 9 0 0 0 NA NA NA NA NA NA NA NA## 10 0 0 0 1 0 8 0 1 1 1 1## INTENSI PP04A PP04B_COD PP04B1 PP04B2 PP04B3_MES PP04B3_ANO PP04B3_DIA PP04C## 1 1 2 4000 2 0 0 0 0 1## 2 NA NA NA NA NA NA NA NA NA## 3 NA NA NA NA NA NA NA NA NA## 4 NA NA NA NA NA NA NA NA NA## 5 NA NA NA NA NA NA NA NA NA## 6 NA NA NA NA NA NA NA NA NA## 7 1 2 4000 2 0 0 0 0 2## 8 NA NA NA NA NA NA NA NA NA## 9 NA NA NA NA NA NA NA NA NA## 10 1 2 9700 1 1 0 0 2 0## PP04C99 PP04D_COD PP04G PP05B2_MES PP05B2_ANO PP05B2_DIA PP05C_1 PP05C_2## 1 0 72113 5 0 0 0 1 3## 2 NA NA NA NA NA NA NA NA## 3 NA NA NA NA NA NA NA NA## 4 NA NA NA NA NA NA NA NA## 5 NA NA NA NA NA NA NA NA## 6 NA NA NA NA NA NA NA NA## 7 0 72314 8 0 0 0 0 0## 8 NA NA NA NA NA NA NA NA## 9 NA NA NA NA NA NA NA NA## 10 0 55314 0 0 0 0 0 0## PP05C_3 PP05E PP05F PP05H PP06A PP06C PP06D PP06E PP06H PP07A PP07C PP07D## 1 3 1 7 6 2 20000 0 0 0 0 0 0## 2 NA NA NA NA NA NA NA NA NA NA NA NA## 3 NA NA NA NA NA NA NA NA NA NA NA NA## 4 NA NA NA NA NA NA NA NA NA NA NA NA## 5 NA NA NA NA NA NA NA NA NA NA NA NA## 6 NA NA NA NA NA NA NA NA NA NA NA NA## 7 0 0 0 0 0 0 0 0 0 1 1 1## 8 NA NA NA NA NA NA NA NA NA NA NA NA## 9 NA NA NA NA NA NA NA NA NA NA NA NA## 10 0 0 0 0 0 0 0 0 0 0 0 0## PP07E PP07F1 PP07F2 PP07F3 PP07F4 PP07F5 PP07G1 PP07G2 PP07G3 PP07G4## 1 0 0 0 0 0 0 0 0 0 0## 2 NA NA NA NA NA NA NA NA NA NA## 3 NA NA NA NA NA NA NA NA NA NA## 4 NA NA NA NA NA NA NA NA NA NA## 5 NA NA NA NA NA NA NA NA NA NA## 6 NA NA NA NA NA NA NA NA NA NA## 7 4 2 2 2 2 5 2 2 2 2## 8 NA NA NA NA NA NA NA NA NA NA## 9 NA NA NA NA NA NA NA NA NA NA## 10 0 2 2 2 2 5 2 2 2 2## PP07G_59 PP07H PP07I PP07J PP07K PP08D1 PP08D4 PP08F1 PP08F2 PP08J1 PP08J2## 1 0 0 0 0 0 0 0 0 0 0 0## 2 NA NA NA NA NA NA NA NA NA NA NA## 3 NA NA NA NA NA NA NA NA NA NA NA## 4 NA NA NA NA NA NA NA NA NA NA NA## 5 NA NA NA NA NA NA NA NA NA NA NA## 6 NA NA NA NA NA NA NA NA NA NA NA## 7 5 2 2 1 4 0 0 0 0 0 0## 8 NA NA NA NA NA NA NA NA NA NA NA## 9 NA NA NA NA NA NA NA NA NA NA NA## 10 5 2 2 1 4 0 0 0 0 0 0## PP08J3 PP09A PP09A_ESP PP09B PP09C PP09C_ESP PP10A PP10C PP10D PP10E PP11A## 1 0 0 0 0 NA NA NA NA NA## 2 NA NA NA NA NA NA NA NA NA## 3 NA NA NA NA NA NA NA NA NA## 4 NA NA NA NA NA NA NA NA NA## 5 NA NA NA NA NA NA NA NA NA## 6 NA NA NA NA NA NA NA NA NA## 7 0 0 0 0 NA NA NA NA NA## 8 NA NA NA NA NA NA NA NA NA## 9 NA NA NA NA NA NA NA NA NA## 10 0 0 0 0 NA NA NA NA NA## PP11B_COD PP11B1 PP11B2_MES PP11B2_ANO PP11B2_DIA PP11C PP11C99 PP11D_COD## 1 NA NA NA NA NA NA NA NA## 2 NA NA NA NA NA NA NA NA## 3 NA NA NA NA NA NA NA NA## 4 NA NA NA NA NA NA NA NA## 5 NA NA NA NA NA NA NA NA## 6 NA NA NA NA NA NA NA NA## 7 NA NA NA NA NA NA NA NA## 8 NA NA NA NA NA NA NA NA## 9 NA NA NA NA NA NA NA NA## 10 NA NA NA NA NA NA NA NA## PP11G_ANO PP11G_MES PP11G_DIA PP11L PP11L1 PP11M PP11N PP11O PP11P PP11Q## 1 NA NA NA NA NA NA NA NA NA NA## 2 NA NA NA NA NA NA NA NA NA NA## 3 NA NA NA NA NA NA NA NA NA NA## 4 NA NA NA NA NA NA NA NA NA NA## 5 NA NA NA NA NA NA NA NA NA NA## 6 NA NA NA NA NA NA NA NA NA NA## 7 NA NA NA NA NA NA NA NA NA NA## 8 NA NA NA NA NA NA NA NA NA NA## 9 NA NA NA NA NA NA NA NA NA NA## 10 NA NA NA NA NA NA NA NA NA NA## PP11R PP11S PP11T P21 DECOCUR IDECOCUR RDECOCUR GDECOCUR PDECOCUR ADECOCUR## 1 NA NA NA 20000 1 2 2 1 NA 2## 2 NA NA NA 0 0 0 0 0 NA 0## 3 NA NA NA 0 0 0 0 0 NA 0## 4 NA NA NA 0 0 NA 0 0 NA 0## 5 NA NA NA 0 0 NA 0 0 NA 0## 6 NA NA NA 0 0 0 0 0 NA 0## 7 NA NA NA 0 0 0 0 0 NA 0## 8 NA NA NA 0 0 0 0 0 NA 0## 9 NA NA NA 0 0 0 0 0 NA 0## 10 NA NA NA 0 0 0 0 0 NA 0## PONDIIO TOT_P12 P47T DECINDR IDECINDR RDECINDR GDECINDR PDECINDR ADECINDR## 1 580 0 46000 4 4 4 4 NA 4## 2 543 0 44400 4 4 4 4 NA 4## 3 543 0 276000 10 10 10 10 NA 10## 4 894 0 -9 12 NA 12 12 NA 12## 5 894 0 -9 12 NA 12 12 NA 12## 6 345 0 5000 1 1 1 1 NA 1## 7 345 15000 15000 1 1 1 1 NA 2## 8 345 0 0 0 0 0 0 NA 0## 9 345 0 31800 2 3 3 2 NA 3## 10 345 0 0 0 0 0 0 NA 0## PONDII V2_M V3_M V4_M V5_M V8_M V9_M V10_M V11_M V12_M V18_M V19_AM V21_M## 1 565 26000 0 0 0 0 0 0 0 0 0 0 0## 2 561 44400 0 0 0 0 0 0 0 0 0 0 0## 3 567 276000 0 0 0 0 0 0 0 0 0 0 0## 4 0 -9 0 0 0 0 0 0 0 0 0 0 0## 5 0 -9 0 0 0 0 0 0 0 0 0 0 0## 6 363 0 0 0 5000 0 0 0 0 0 0 0 0## 7 394 0 0 0 0 0 0 0 0 0 0 0 0## 8 345 0 0 0 0 0 0 0 0 0 0 0 0## 9 345 31800 0 0 0 0 0 0 0 0 0 0 0## 10 345 0 0 0 0 0 0 0 0 0 0 0 0## T_VI ITF DECIFR IDECIFR RDECIFR GDECIFR PDECIFR ADECIFR IPCF DECCFR## 1 26000 46000 2 1 1 2 NA 1 46000 5## 2 44400 320400 10 10 10 10 NA 10 160200 10## 3 276000 320400 10 10 10 10 NA 10 160200 10## 4 -9 0 12 NA 12 12 NA 12 0 12## 5 -9 0 12 NA 12 12 NA 12 0 12## 6 5000 51800 2 2 2 2 NA 2 10360 1## 7 0 51800 2 2 2 2 NA 2 10360 1## 8 0 51800 2 2 2 2 NA 2 10360 1## 9 31800 51800 2 2 2 2 NA 2 10360 1## 10 0 51800 2 2 2 2 NA 2 10360 1## IDECCFR RDECCFR GDECCFR PDECCFR ADECCFR PONDIH## 1 6 6 5 NA 5 474## 2 10 10 10 NA 10 611## 3 10 10 10 NA 10 611## 4 NA 12 12 NA 12 0## 5 NA 12 12 NA 12 0## 6 1 1 1 NA 1 321## 7 1 1 1 NA 1 321## 8 1 1 1 NA 1 321## 9 1 1 1 NA 1 321## 10 1 1 1 NA 1 321Explorar la base de datos
Para visualizar la base completa la clickeamos yendo al entorno o sino corremos
View(eph_ind)Y se abre una pestaña que muestra el dataframe completo, similar a esto
| CODUSU | ANO4 | TRIMESTRE | NRO_HOGAR | COMPONENTE | H15 | REGION | MAS_500 | AGLOMERADO | PONDERA | CH03 | CH04 | CH05 | CH06 | CH07 | CH08 | CH09 | CH10 | CH11 | CH12 | CH13 | CH14 | CH15 | CH15_COD | CH16 | CH16_COD | NIVEL_ED | ESTADO | CAT_OCUP | CAT_INAC |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| TQRMNORVQHJNLTCDEGGFB00788741 | 2022 | 3 | 1 | 1 | 1 | 42 | S | 10 | 537 | 1 | 1 | 10/04/1961 | 61 | 5 | 3 | 1 | 2 | 0 | 4 | 2 | 2 | 1 | NA | 1 | NA | 3 | 1 | 2 | 0 |
| TQRMNOQSWHKNKRCDEGJBF00789123 | 2022 | 3 | 1 | 1 | 1 | 43 | S | 13 | 543 | 1 | 1 | 06/10/1953 | 68 | 2 | 1 | 1 | 2 | 0 | 4 | 1 | NA | 1 | NA | 1 | NA | 4 | 3 | 0 | 1 |
| TQRMNOQSWHKNKRCDEGJBF00789123 | 2022 | 3 | 1 | 2 | 1 | 43 | S | 13 | 543 | 2 | 2 | 28/08/1955 | 66 | 2 | 1 | 1 | 2 | 0 | 7 | 1 | NA | 1 | NA | 1 | NA | 6 | 3 | 0 | 1 |
| TQRMNOTSQHLLMLCDEIIAD00703213 | 2022 | 3 | 1 | 1 | 1 | 1 | S | 32 | 894 | 1 | 1 | 10/03/1957 | 65 | 2 | 12 | 1 | 2 | 0 | 8 | 1 | NA | 1 | NA | 1 | NA | 6 | 3 | 0 | 1 |
| TQRMNOTSQHLLMLCDEIIAD00703213 | 2022 | 3 | 1 | 2 | 1 | 1 | S | 32 | 894 | 2 | 2 | 07/01/1958 | 64 | 2 | 12 | 1 | 2 | 0 | 6 | 1 | NA | 1 | NA | 1 | NA | 6 | 3 | 0 | 1 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 2022 | 3 | 1 | 1 | 1 | 40 | S | 29 | 345 | 1 | 2 | 18/09/1977 | 44 | 5 | 4 | 1 | 2 | 0 | 2 | 2 | 4 | 1 | NA | 1 | NA | 1 | 3 | 0 | 4 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 2022 | 3 | 1 | 2 | 1 | 40 | S | 29 | 345 | 3 | 1 | 13/05/2003 | 19 | 1 | 4 | 1 | 2 | 0 | 4 | 2 | 1 | 1 | NA | 1 | NA | 3 | 1 | 3 | 0 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 2022 | 3 | 1 | 3 | 1 | 40 | S | 29 | 345 | 3 | 1 | 30/03/2010 | 12 | 5 | 4 | 1 | 1 | 1 | 4 | 2 | 0 | 1 | NA | 1 | NA | 3 | 3 | 0 | 3 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 2022 | 3 | 1 | 4 | 0 | 40 | S | 29 | 345 | 3 | 1 | 06/03/2015 | 7 | 5 | 3 | 2 | 1 | 1 | 2 | 2 | 1 | 1 | NA | 1 | NA | 1 | 4 | 0 | 1 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 2022 | 3 | 1 | 5 | 1 | 40 | S | 29 | 345 | 4 | 2 | 30/04/2005 | 17 | 1 | 4 | 1 | 2 | 0 | 4 | 2 | 1 | 1 | NA | 1 | NA | 3 | 1 | 3 | 0 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 2022 | 3 | 1 | 1 | 1 | 44 | N | 91 | 141 | 1 | 1 | 17/08/1970 | 52 | 2 | 1 | 1 | 2 | 0 | 4 | 1 | NA | 1 | NA | 1 | NA | 4 | 2 | 0 | 0 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 2022 | 3 | 1 | 2 | 1 | 44 | N | 91 | 141 | 2 | 2 | 03/07/1974 | 48 | 2 | 1 | 1 | 2 | 0 | 7 | 1 | NA | 1 | NA | 1 | NA | 6 | 1 | 3 | 0 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 2022 | 3 | 1 | 3 | 1 | 44 | N | 91 | 141 | 3 | 1 | 16/08/2007 | 15 | 5 | 1 | 1 | 1 | 1 | 4 | 2 | 2 | 1 | NA | 1 | NA | 3 | 3 | 0 | 3 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 2022 | 3 | 1 | 4 | 1 | 44 | N | 91 | 141 | 3 | 1 | 17/09/2010 | 11 | 5 | 1 | 1 | 1 | 1 | 2 | 2 | 5 | 1 | NA | 1 | NA | 1 | 3 | 0 | 3 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 2022 | 3 | 1 | 1 | 1 | 41 | N | 7 | 303 | 1 | 1 | 19/08/1968 | 54 | 1 | 1 | 1 | 2 | 0 | 2 | 2 | 3 | 4 | 221 | 1 | NA | 1 | 1 | 3 | 0 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 2022 | 3 | 1 | 2 | 1 | 41 | N | 7 | 303 | 2 | 2 | 28/05/1970 | 52 | 1 | 1 | 1 | 2 | 0 | 2 | 2 | 3 | 4 | 221 | 1 | NA | 1 | 3 | 0 | 7 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 2022 | 3 | 1 | 3 | 1 | 41 | N | 7 | 303 | 3 | 2 | 09/01/1997 | 25 | 5 | 4 | 1 | 1 | 1 | 4 | 2 | 1 | 1 | NA | 1 | NA | 3 | 1 | 3 | 0 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 2022 | 3 | 1 | 4 | 1 | 41 | N | 7 | 303 | 3 | 2 | 08/11/2003 | 18 | 5 | 1 | 1 | 2 | 0 | 4 | 2 | 1 | 1 | NA | 1 | NA | 3 | 3 | 0 | 7 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 2022 | 3 | 1 | 5 | 0 | 41 | N | 7 | 303 | 5 | 1 | 12/05/2014 | 8 | 5 | 4 | 1 | 1 | 1 | 2 | 2 | 2 | 1 | NA | 1 | NA | 1 | 4 | 0 | 3 |
| TQRMNOPXUHLMKUCDEGOIH00706727 | 2022 | 3 | 1 | 1 | 1 | 40 | N | 18 | 275 | 1 | 1 | 03/03/1983 | 39 | 2 | 1 | 1 | 2 | 0 | 4 | 1 | NA | 3 | 54 | 1 | NA | 4 | 1 | 3 | 0 |
| TQRMNOPXUHLMKUCDEGOIH00706727 | 2022 | 3 | 1 | 2 | 1 | 40 | N | 18 | 275 | 2 | 2 | 26/08/1985 | 37 | 2 | 1 | 1 | 2 | 0 | 4 | 1 | NA | 3 | 54 | 1 | NA | 4 | 3 | 0 | 4 |
| TQRMNOPXUHLMKUCDEGOIH00706727 | 2022 | 3 | 1 | 3 | 1 | 40 | N | 18 | 275 | 3 | 1 | 04/02/2012 | 10 | 5 | 1 | 1 | 1 | 1 | 2 | 2 | 3 | 3 | 54 | 1 | NA | 1 | 3 | 0 | 3 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 2022 | 3 | 1 | 1 | 1 | 41 | N | 12 | 333 | 1 | 1 | 20/12/1994 | 27 | 1 | 1 | 1 | 2 | 0 | 6 | 1 | NA | 3 | 94 | 1 | NA | 6 | 1 | 3 | 0 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 2022 | 3 | 1 | 2 | 1 | 41 | N | 12 | 333 | 2 | 2 | 29/11/1993 | 28 | 1 | 1 | 1 | 2 | 0 | 6 | 2 | 3 | 1 | NA | 1 | NA | 5 | 1 | 3 | 0 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 2022 | 3 | 1 | 3 | 0 | 41 | N | 12 | 333 | 3 | 2 | 28/11/2014 | 7 | 5 | 1 | 1 | 1 | 1 | 2 | 2 | 1 | 1 | NA | 1 | NA | 1 | 4 | 0 | 3 |
(aquí mostramos solo las primeras 50 filas y 30 columnas)
Manipular datos
Ahora trabajemos sobre los datos que cargamos.
Manipular datos
Ahora trabajemos sobre los datos que cargamos.
Apliquemos lo que aprendimos y filtremos la base, dejando solo los individuos del aglomerado 18 (Resistencia - Chaco), que no tengan ingreso missing y que sean mujeres
Manipular datos
Ahora trabajemos sobre los datos que cargamos.
Apliquemos lo que aprendimos y filtremos la base, dejando solo los individuos del aglomerado 18 (Resistencia - Chaco), que no tengan ingreso missing y que sean mujeres
eph_ind[eph_ind$AGLOMERADO==18 & !is.na(eph_ind$IPCF) & eph_ind$CH04==2,]Manipular datos
Si hacemos esto mismo pero llamndo solo las columnas que nos interesan esas columnas deberían tener los valores de la condición que definen este subgrupo.
eph_ind[eph_ind$AGLOMERADO==18 & !is.na(eph_ind$IPCF) & eph_ind$CH04==2, c("AGLOMERADO", "IPCF", "CH04")]## AGLOMERADO IPCF CH04## 21 18 26666.67 2## 119 18 20500.00 2## 125 18 31540.00 2## 126 18 31540.00 2## 128 18 31540.00 2## 146 18 39357.14 2## 147 18 39357.14 2## 151 18 39357.14 2## 291 18 11300.00 2## 292 18 11300.00 2## 294 18 11300.00 2## 440 18 11666.67 2## 441 18 11666.67 2## 444 18 11666.67 2## 464 18 29083.33 2## 466 18 29083.33 2## 468 18 29083.33 2## 469 18 29083.33 2## 983 18 8850.00 2## 985 18 8850.00 2## 986 18 8850.00 2## 997 18 31583.33 2## 999 18 31583.33 2## 1000 18 31583.33 2## 1487 18 35900.00 2## 1492 18 35900.00 2## 1493 18 35900.00 2## 1549 18 35900.00 2## 1630 18 45000.00 2## 1766 18 64700.00 2## 1977 18 35000.00 2## 2138 18 14750.00 2## 2139 18 110000.00 2## 2145 18 25000.00 2## 2147 18 25000.00 2## 2360 18 0.00 2## 2379 18 30000.00 2## 2420 18 24250.00 2## 2506 18 23333.33 2## 2508 18 23333.33 2## 2957 18 19258.33 2## 2959 18 19258.33 2## 2960 18 19258.33 2## 2961 18 19258.33 2## 3037 18 9400.00 2## 3074 18 100833.33 2## 3077 18 30140.00 2## 3079 18 30140.00 2## 3080 18 30140.00 2## 3081 18 27200.00 2## 3137 18 27200.00 2## 3138 18 27200.00 2## 3169 18 57500.00 2## 3660 18 24900.00 2## 3703 18 27600.00 2## 3704 18 27600.00 2## 3705 18 27600.00 2## 3735 18 76000.00 2## 3736 18 76000.00 2## 4248 18 20050.00 2## 4249 18 20050.00 2## 4256 18 33566.67 2## 4258 18 33566.67 2## 4260 18 17600.00 2## 4261 18 17600.00 2## 4314 18 17600.00 2## 4315 18 17600.00 2## 4418 18 43250.00 2## 4419 18 43250.00 2## 4421 18 43250.00 2## 4477 18 20225.00 2## 4633 18 26666.67 2## 4634 18 26666.67 2## 4635 18 26666.67 2## 4804 18 102000.00 2## 4899 18 38625.00 2## 4900 18 38625.00 2## 4955 18 38625.00 2## 4962 18 37833.33 2## 4963 18 37833.33 2## 4964 18 37833.33 2## 4965 18 37833.33 2## 5035 18 80000.00 2## 5526 18 77000.00 2## 5567 18 24454.55 2## 5615 18 70000.00 2## 5622 18 24325.00 2## 5626 18 24454.55 2## 5627 18 24454.55 2## 5668 18 55375.00 2## 5670 18 55375.00 2## 5677 18 24325.00 2## 5739 18 36750.00 2## 5740 18 36750.00 2## 5741 18 36750.00 2## 5753 18 83333.33 2## 5754 18 83333.33 2## 6022 18 28000.00 2## 6024 18 28000.00 2## 6026 18 28000.00 2## 6509 18 22750.00 2## 6620 18 64666.67 2## 6634 18 20000.00 2## 6701 18 57500.00 2## 6830 18 0.00 2## 6831 18 0.00 2## 6832 18 0.00 2## 6836 18 0.00 2## 6837 18 0.00 2## 7178 18 52500.00 2## 7188 18 25900.00 2## 7189 18 25900.00 2## 7190 18 25900.00 2## 7205 18 100000.00 2## 7318 18 20000.00 2## 7320 18 20000.00 2## 7345 18 17750.00 2## 7346 18 17750.00 2## 7605 18 23975.00 2## 7608 18 23975.00 2## 7610 18 108000.00 2## 7611 18 108000.00 2## 7746 18 30000.00 2## 7747 18 30000.00 2## 7748 18 30000.00 2## 7765 18 52500.00 2## 7884 18 108000.00 2## 7886 18 23750.00 2## 7887 18 23750.00 2## 7967 18 52500.00 2## 7969 18 52500.00 2## 8245 18 35666.67 2## 8493 18 27000.00 2## 8537 18 30000.00 2## 8538 18 30000.00 2## 8826 18 38000.00 2## 8832 18 26000.00 2## 8834 18 26000.00 2## 8841 18 82500.00 2## 8845 18 20050.00 2## 8937 18 12250.00 2## 8938 18 12250.00 2## 9125 18 21000.00 2## 9127 18 21000.00 2## 9221 18 28333.33 2## 9222 18 28333.33 2## 9227 18 20000.00 2## 9229 18 20000.00 2## 9231 18 20000.00 2## 9233 18 28000.00 2## 9235 18 28000.00 2## 10109 18 14950.00 2## 10110 18 14950.00 2## 10215 18 32300.00 2## 10435 18 26333.33 2## 10436 18 26333.33 2## 10635 18 90000.00 2## 10661 18 15000.00 2## 10800 18 29400.00 2## 10819 18 29900.00 2## 10822 18 29900.00 2## 10823 18 29900.00 2## 11029 18 90000.00 2## 11116 18 90000.00 2## 11117 18 90000.00 2## 11141 18 10833.33 2## 11144 18 10833.33 2## 11145 18 10833.33 2## 11146 18 10833.33 2## 11224 18 39375.00 2## 11315 18 39375.00 2## 11406 18 137500.00 2## 11407 18 137500.00 2## 11463 18 31866.67 2## 11603 18 43125.00 2## 11604 18 43125.00 2## 11834 18 55000.00 2## 11853 18 33750.00 2## 12157 18 60375.00 2## 12159 18 60375.00 2## 12277 18 49500.00 2## 12310 18 96000.00 2## 12366 18 25571.43 2## 12370 18 25571.43 2## 12371 18 25571.43 2## 12372 18 25571.43 2## 12699 18 30750.00 2## 12722 18 9500.00 2## 12723 18 9500.00 2## 12725 18 9500.00 2## 12903 18 23625.00 2## 12904 18 23625.00 2## 12905 18 23625.00 2## 13039 18 22112.50 2## 13041 18 22112.50 2## 13042 18 22112.50 2## 13064 18 83500.00 2## 13173 18 70000.00 2## 13174 18 70000.00 2## 13236 18 21600.00 2## 13238 18 21600.00 2## 13308 18 150000.00 2## 13401 18 3400.00 2## 13461 18 18690.00 2## 13463 18 18690.00 2## 13516 18 35500.00 2## 13518 18 35500.00 2## 13734 18 18690.00 2## 13737 18 18690.00 2## 13850 18 27500.00 2## 13851 18 27500.00 2## 13852 18 27500.00 2## 13916 18 0.00 2## 13917 18 0.00 2## 13918 18 0.00 2## 13919 18 0.00 2## 14127 18 61875.00 2## 14129 18 61875.00 2## 14186 18 61666.67 2## 14188 18 61666.67 2## 14236 18 37750.00 2## 14265 18 52500.00 2## 14287 18 26166.67 2## 14289 18 26166.67 2## 14291 18 26166.67 2## 14346 18 58125.00 2## 14348 18 58125.00 2## 14574 18 234166.67 2## 14649 18 16400.00 2## 14650 18 16400.00 2## 14651 18 16400.00 2## 14652 18 16400.00 2## 14653 18 16400.00 2## 14744 18 54500.00 2## 14745 18 54500.00 2## 14746 18 54500.00 2## 14821 18 47600.00 2## 14904 18 30000.00 2## 14905 18 30000.00 2## 14906 18 30000.00 2## 15075 18 47875.00 2## 15076 18 47875.00 2## 15078 18 47875.00 2## 15086 18 117500.00 2## 15172 18 89000.00 2## 15316 18 16250.00 2## 15445 18 21425.00 2## 15447 18 21425.00 2## 15585 18 16250.00 2## 15951 18 14833.33 2## 15952 18 14833.33 2## 15953 18 14833.33 2## 16112 18 24666.67 2## 16228 18 13583.33 2## 16230 18 13583.33 2## 16231 18 13583.33 2## 16232 18 13583.33 2## 16234 18 49500.00 2## 16499 18 36750.00 2## 16501 18 36750.00 2## 16502 18 82416.67 2## 16551 18 17833.33 2## 16552 18 17833.33 2## 16553 18 17833.33 2## 16555 18 65000.00 2## 16565 18 33666.67 2## 16854 18 34900.00 2## 16856 18 34900.00 2## 16858 18 34900.00 2## 16860 18 12000.00 2## 16861 18 12000.00 2## 16862 18 12000.00 2## 16863 18 12000.00 2## 17045 18 63000.00 2## 17083 18 37950.00 2## 17084 18 37950.00 2## 17112 18 41000.00 2## 17289 18 17333.33 2## 17291 18 17333.33 2## 17327 18 24825.00 2## 17328 18 24825.00 2## 17531 18 34000.00 2## 17532 18 34000.00 2## 17533 18 34000.00 2## 17534 18 34000.00 2## 17548 18 31983.33 2## 17549 18 31983.33 2## 17553 18 31983.33 2## 17601 18 22750.00 2## 17642 18 79000.00 2## 17643 18 79000.00 2## 17648 18 55500.00 2## 17771 18 17000.00 2## 17773 18 17000.00 2## 17789 18 70583.33 2## 17790 18 70583.33 2## 17929 18 26833.33 2## 18036 18 33833.33 2## 18037 18 33833.33 2## 18082 18 48750.00 2## 18135 18 26000.00 2## 18149 18 41666.67 2## 18204 18 30000.00 2## 18205 18 30000.00 2## 18223 18 0.00 2## 18227 18 11625.00 2## 18404 18 26000.00 2## 18405 18 26000.00 2## 18413 18 38333.33 2## 18633 18 44166.67 2## 18634 18 44166.67 2## 18645 18 46500.00 2## 18646 18 46500.00 2## 18648 18 46500.00 2## 18797 18 60000.00 2## 18798 18 60000.00 2## 18840 18 27500.00 2## 18843 18 27500.00 2## 19064 18 33800.00 2## 19065 18 33800.00 2## 19071 18 45833.33 2## 19074 18 45833.33 2## 19128 18 75000.00 2## 19317 18 18000.00 2## 19320 18 18000.00 2## 19321 18 18000.00 2## 19322 18 18000.00 2## 19481 18 120000.00 2## 19503 18 45000.00 2## 19504 18 45000.00 2## 19707 18 72500.00 2## 20016 18 17875.00 2## 20017 18 17875.00 2## 20077 18 17300.00 2## 20151 18 41250.00 2## 20152 18 41250.00 2## 20303 18 31250.00 2## 20306 18 31250.00 2## 20308 18 31250.00 2## 20309 18 31250.00 2## 20347 18 17300.00 2## 20348 18 17300.00 2## 20353 18 36500.00 2## 20361 18 55500.00 2## 20756 18 112333.33 2## 20770 18 131000.00 2## 20779 18 17850.00 2## 20781 18 17850.00 2## 20784 18 17850.00 2## 20785 18 17850.00 2## 20787 18 17850.00 2## 21176 18 6900.00 2## 21179 18 6900.00 2## 21180 18 6900.00 2## 21439 18 33500.00 2## 21440 18 33500.00 2## 21441 18 33500.00 2## 21524 18 25000.00 2## 21525 18 25000.00 2## 21791 18 40000.00 2## 21793 18 52333.33 2## 21926 18 97500.00 2## 21947 18 35000.00 2## 21948 18 35000.00 2## 22098 18 18280.00 2## 22099 18 18280.00 2## 22100 18 18280.00 2## 22101 18 18280.00 2## 22676 18 17333.33 2## 22761 18 30000.00 2## 23630 18 26333.33 2## 23631 18 26333.33 2## 23775 18 0.00 2## 23777 18 0.00 2## 24205 18 13983.33 2## 24206 18 13983.33 2## 24209 18 13983.33 2## 24210 18 13983.33 2## 24321 18 37333.33 2## 24322 18 37333.33 2## 24323 18 37333.33 2## 24579 18 68500.00 2## 24635 18 19000.00 2## 24636 18 19000.00 2## 24638 18 19000.00 2## 24753 18 36000.00 2## 24924 18 14000.00 2## 24980 18 70000.00 2## 25030 18 44000.00 2## 25167 18 24333.33 2## 25169 18 24333.33 2## 25387 18 11500.00 2## 25389 18 11500.00 2## 25407 18 27812.50 2## 25409 18 27812.50 2## 25498 18 187500.00 2## 25684 18 27812.50 2## 25794 18 15000.00 2## 25796 18 11800.00 2## 25797 18 11800.00 2## 25798 18 11800.00 2## 25799 18 11800.00 2## 25926 18 19166.67 2## 25929 18 19166.67 2## 26042 18 43750.00 2## 26043 18 43750.00 2## 26044 18 43750.00 2## 26051 18 20000.00 2## 26286 18 15000.00 2## 26288 18 15000.00 2## 26289 18 15000.00 2## 26317 18 92000.00 2## 26324 18 16250.00 2## 26601 18 39000.00 2## 26603 18 39000.00 2## 26614 18 62900.00 2## 26720 18 35000.00 2## 26722 18 35000.00 2## 26747 18 26583.33 2## 26750 18 26583.33 2## 26759 18 21600.00 2## 26855 18 11877.78 2## 26857 18 11877.78 2## 26859 18 11877.78 2## 26861 18 11877.78 2## 26862 18 11877.78 2## 27013 18 23928.57 2## 27016 18 23928.57 2## 27147 18 50150.00 2## 27149 18 50150.00 2## 27160 18 56900.00 2## 27162 18 56900.00 2## 27163 18 56900.00 2## 27169 18 26750.00 2## 27248 18 16800.00 2## 27249 18 16800.00 2## 27250 18 16800.00 2## 27251 18 16800.00 2## 27264 18 20666.67 2## 27265 18 20666.67 2## 27435 18 26750.00 2## 27542 18 11900.00 2## 27543 18 11900.00 2## 27545 18 11900.00 2## 27579 18 17500.00 2## 27580 18 17500.00 2## 27581 18 17500.00 2## 27582 18 17500.00 2## 27583 18 17500.00 2## 27893 18 20000.00 2## 27894 18 20000.00 2## 27919 18 11666.67 2## 27920 18 11666.67 2## 27922 18 11666.67 2## 28158 18 33333.33 2## 28163 18 0.00 2## 28199 18 56500.00 2## 28200 18 56500.00 2## 28316 18 17750.00 2## 28317 18 17750.00 2## 28318 18 17750.00 2## 28433 18 0.00 2## 28565 18 17400.00 2## 28581 18 21100.00 2## 28583 18 21100.00 2## 28585 18 21100.00 2## 28658 18 12500.00 2## 28659 18 12500.00 2## 28696 18 11600.00 2## 28698 18 11600.00 2## 28699 18 11600.00 2## 28816 18 39000.00 2## 29052 18 65000.00 2## 29053 18 65000.00 2## 29219 18 20000.00 2## 29221 18 20000.00 2## 29222 18 20000.00 2## 29285 18 27750.00 2## 29366 18 25125.00 2## 29367 18 25125.00 2## 29481 18 12900.00 2## 29484 18 12900.00 2## 29511 18 17833.33 2## 29544 18 48500.00 2## 29549 18 23060.00 2## 29550 18 23060.00 2## 29553 18 23060.00 2## 29606 18 0.00 2## 29638 18 25125.00 2## 29679 18 45000.00 2## 29690 18 23333.33 2## 29785 18 17833.33 2## 29786 18 17833.33 2## 29787 18 12262.50 2## 29792 18 12262.50 2## 29796 18 60000.00 2## 29993 18 151000.00 2## 30059 18 35000.00 2## 30060 18 35000.00 2## 30143 18 40000.00 2## 30144 18 40000.00 2## 30184 18 13250.00 2## 30185 18 13250.00 2## 30204 18 22100.00 2## 30205 18 22100.00 2## 30207 18 22100.00 2## 30215 18 40333.33 2## 30216 18 40333.33 2## 30470 18 17420.00 2## 30473 18 17420.00 2## 30544 18 19100.00 2## 30545 18 19100.00 2## 30546 18 19100.00 2## 30604 18 19100.00 2## 30607 18 19100.00 2## 30613 18 33000.00 2## 30614 18 33000.00 2## 30633 18 35625.00 2## 30789 18 68050.00 2## 30790 18 68050.00 2## 30791 18 68050.00 2## 31007 18 16666.67 2## 31210 18 36000.00 2## 31212 18 25450.00 2## 31232 18 13666.67 2## 31233 18 13666.67 2## 31234 18 13666.67 2## 31243 18 16200.00 2## 31245 18 16200.00 2## 31458 18 9750.00 2## 31459 18 9750.00 2## 31461 18 9750.00 2## 31485 18 13000.00 2## 31487 18 13000.00 2## 31492 18 35750.00 2## 31615 18 45966.67 2## 32001 18 17000.00 2## 32270 18 17000.00 2## 32420 18 13214.29 2## 32425 18 13214.29 2## 32426 18 13214.29 2## 32529 18 24325.00 2## 32531 18 24325.00 2## 32532 18 24325.00 2## 32703 18 25333.33 2## 32717 18 24125.00 2## 32718 18 24125.00 2## 32894 18 4875.00 2## 32895 18 4875.00 2## 32896 18 4875.00 2## 32897 18 4875.00 2## 32992 18 11200.00 2## 32994 18 11200.00 2## 32996 18 11200.00 2## 33271 18 13400.00 2## 33272 18 13400.00 2## 33542 18 13400.00 2## 33544 18 13400.00 2## 33708 18 36250.00 2## 33709 18 36250.00 2## 33710 18 36250.00 2## 33714 18 34000.00 2## 33716 18 34000.00 2## 33717 18 43000.00 2## 33820 18 75000.00 2## 33821 18 75000.00 2## 33864 18 35250.00 2## 33865 18 35250.00 2## 33867 18 35250.00 2## 33987 18 43000.00 2## 34163 18 13333.33 2## 34199 18 138333.33 2## 34220 18 14833.33 2## 34234 18 19875.00 2## 34235 18 35000.00 2## 34238 18 97500.00 2## 34302 18 30966.67 2## 34436 18 42500.00 2## 34438 18 42500.00 2## 34501 18 19875.00 2## 34573 18 50000.00 2## 34717 18 20166.67 2## 34718 18 20166.67 2## 34720 18 20166.67 2## 34721 18 20166.67 2## 34770 18 42666.67 2## 34771 18 42666.67 2## 34772 18 42666.67 2## 34823 18 41000.00 2## 34824 18 41000.00 2## 34871 18 51000.00 2## 34993 18 22000.00 2## 34996 18 22000.00 2## 34997 18 22000.00 2## 35474 18 52000.00 2## 35475 18 52000.00 2## 35556 18 36746.67 2## 35557 18 36746.67 2## 35566 18 98000.00 2## 35624 18 22500.00 2## 35625 18 22500.00 2## 35788 18 31500.00 2## 35806 18 28000.00 2## 35877 18 33000.00 2## 35878 18 33000.00 2## 35879 18 33000.00 2## 35891 18 90000.00 2## 35901 18 45000.00 2## 36010 18 26133.33 2## 36015 18 26133.33 2## 36016 18 26133.33 2## 36020 18 42675.00 2## 36058 18 31500.00 2## 36075 18 28000.00 2## 36286 18 42675.00 2## 36336 18 17600.00 2## 36338 18 17600.00 2## 36352 18 137000.00 2## 36496 18 23900.00 2## 36512 18 21333.33 2## 36594 18 27700.00 2## 36596 18 27700.00 2## 36598 18 27700.00 2## 36608 18 17600.00 2## 36676 18 18250.00 2## 36683 18 35000.00 2## 36684 18 35000.00 2## 36901 18 25333.33 2## 36913 18 31666.67 2## 36914 18 31666.67 2## 36920 18 30000.00 2## 36946 18 18250.00 2## 36998 18 37800.00 2## 37054 18 21000.00 2## 37055 18 21000.00 2## 37165 18 30500.00 2## 37168 18 30500.00 2## 37251 18 11833.33 2## 37254 18 45625.00 2## 37255 18 45625.00 2## 37411 18 14250.00 2## 37413 18 14250.00 2## 37422 18 13642.86 2## 37432 18 17250.00 2## 37433 18 17250.00 2## 37434 18 17250.00 2## 37439 18 10166.67 2## 37441 18 10166.67 2## 37442 18 10166.67 2## 37444 18 10166.67 2## 37445 18 10166.67 2## 37465 18 34700.00 2## 37488 18 30000.00 2## 37578 18 35833.33 2## 37579 18 35833.33 2## 37606 18 20720.00 2## 37607 18 20720.00 2## 37609 18 20720.00 2## 37696 18 80000.00 2## 37698 18 80000.00 2## 37711 18 10166.67 2## 37712 18 10166.67 2## 37944 18 85000.00 2## 38026 18 23875.00 2## 38027 18 23875.00 2## 38029 18 23875.00 2## 38216 18 20000.00 2## 38217 18 20000.00 2## 38225 18 58000.00 2## 38303 18 13750.00 2## 38305 18 13750.00 2## 38306 18 13750.00 2## 38499 18 40000.00 2## 38502 18 53000.00 2## 38597 18 62500.00 2## 38655 18 18333.33 2## 38718 18 21360.00 2## 38719 18 21360.00 2## 38721 18 21360.00 2## 38819 18 36000.00 2## 38821 18 36000.00 2## 39006 18 31850.00 2## 39036 18 24516.67 2## 39038 18 24516.67 2## 39039 18 24516.67 2## 39040 18 24516.67 2## 39041 18 24516.67 2## 39172 18 112500.00 2## 39313 18 47500.00 2## 39527 18 66250.00 2## 39529 18 66250.00 2## 39644 18 20083.33 2## 39645 18 20083.33 2## 39648 18 20083.33 2## 39682 18 35000.00 2## 39901 18 22500.00 2## 39903 18 22500.00 2## 39904 18 22500.00 2## 39980 18 44333.33 2## 40052 18 52350.00 2## 40149 18 18500.00 2## 40150 18 18500.00 2## 40151 18 18500.00 2## 40152 18 18500.00 2## 40294 18 40000.00 2## 40296 18 40000.00 2## 40298 18 33766.67 2## 40300 18 33766.67 2## 40302 18 33766.67 2## 40429 18 14666.67 2## 40580 18 26320.00 2## 40581 18 26320.00 2## 40695 18 14666.67 2## 40700 18 29400.00 2## 40702 18 29400.00 2## 40703 18 29400.00 2## 40811 18 16000.00 2## 40892 18 17750.00 2## 40893 18 17750.00 2## 40894 18 17750.00 2## 40895 18 17750.00 2## 41099 18 35000.00 2## 41101 18 35000.00 2## 41440 18 71500.00 2## 41493 18 47500.00 2## 41494 18 47500.00 2## 41495 18 47500.00 2## 41718 18 25250.00 2## 42116 18 36666.67 2## 42117 18 36666.67 2## 42146 18 120000.00 2## 42151 18 22500.00 2## 42157 18 49000.00 2## 42407 18 32500.00 2## 42427 18 49000.00 2## 42491 18 33500.00 2## 42520 18 46375.00 2## 42527 18 25175.00 2## 42530 18 25175.00 2## 42772 18 57900.00 2## 42773 18 57900.00 2## 42827 18 92700.00 2## 43105 18 21880.00 2## 43107 18 21880.00 2## 43211 18 32250.00 2## 43212 18 32250.00 2## 43408 18 17000.00 2## 43409 18 17000.00 2## 43411 18 17000.00 2## 43488 18 16071.43 2## 43489 18 16071.43 2## 43490 18 16071.43 2## 43492 18 16071.43 2## 43493 18 16071.43 2## 43578 18 23333.33 2## 43785 18 13777.78 2## 43789 18 13777.78 2## 43795 18 31000.00 2## 43798 18 31000.00 2## 43823 18 42900.00 2## 43877 18 17520.00 2## 43878 18 17520.00 2## 43909 18 90000.00 2## 44048 18 75000.00 2## 44050 18 75000.00 2## 44058 18 13777.78 2## 44147 18 17520.00 2## 44148 18 17520.00 2## 44157 18 46666.67 2## 44160 18 15000.00 2## 44161 18 15000.00 2## 44237 18 72750.00 2## 44242 18 21425.00 2## 44243 18 21425.00 2## 44244 18 21425.00 2## 44425 18 9233.33 2## 44426 18 9233.33 2## 44428 18 9233.33 2## 44441 18 20833.33 2## 44443 18 20833.33 2## 45115 18 25666.67 2## 45117 18 25666.67 2## 45119 18 25666.67 2## 45123 18 62000.00 2## 45170 18 30580.00 2## 45171 18 30580.00 2## 45173 18 30580.00 2## 45180 18 23000.00 2## 45405 18 45200.00 2## 45407 18 45200.00 2## 45411 18 21250.00 2## 45413 18 21250.00 2## 45414 18 15400.00 2## 45684 18 15400.00 2## 45685 18 15400.00 2## 45755 18 32800.00 2## 45767 18 24500.00 2## 45769 18 24500.00 2## 45914 18 18000.00 2## 45915 18 18000.00 2## 45916 18 18000.00 2## 45957 18 20833.33 2## 45958 18 20833.33 2## 46052 18 24333.33 2## 46053 18 24333.33 2## 46099 18 11000.00 2## 46101 18 11000.00 2## 46243 18 54400.00 2## 46244 18 54400.00 2## 46673 18 15450.00 2## 46675 18 27500.00 2## 46676 18 27500.00 2## 46791 18 36500.00 2## 47032 18 23300.00 2## 47226 18 14400.00 2## 47227 18 14400.00 2## 47229 18 14400.00 2## 47230 18 14400.00 2## 47301 18 23300.00 2## 47515 18 31500.00 2## 47688 18 48000.00 2## 47818 18 46666.67 2## 47843 18 47000.00 2## 47884 18 10000.00 2## 47887 18 10000.00 2## 47888 18 10000.00 2## 48006 18 24450.00 2## 48007 18 24450.00 2## 48066 18 36000.00 2## 48093 18 14600.00 2## 48106 18 68950.00 2## 48116 18 10425.00 2## 48117 18 10425.00 2## 48119 18 10425.00 2## 48120 18 10425.00 2## 48346 18 24775.00 2## 48347 18 24775.00 2## 48360 18 14600.00 2## 48390 18 8924.00 2## 48393 18 8924.00 2## 48394 18 8924.00 2## 48395 18 8924.00 2## 48590 18 38950.00 2## 48592 18 38950.00 2## 48665 18 8924.00 2## 48666 18 8924.00 2## 48732 18 25750.00 2## 48734 18 25750.00 2## 48735 18 25750.00 2## 48833 18 36000.00 2## 48879 18 14400.00 2## 48881 18 14400.00 2## 49000 18 25750.00 2## 49004 18 25750.00 2## 49116 18 26000.00 2## 49118 18 26000.00 2## 49221 18 27200.00 2## 49222 18 27200.00 2De esta forma podemos chequear que filtramos correctamente
Manipular datos
Ahora calculemos el promedio ponderado de edad para este subgrupo con la función weighted.mean()
weighted.mean(eph_ind$CH06[eph_ind$AGLOMERADO==18 & !is.na(eph_ind$IPCF) & eph_ind$CH04==2], na.rm = TRUE)## [1] 34Hicimos un calculo muy simple y ya vemos cómo el código se pone engorroso...
Manipular datos
Ahora calculemos el promedio ponderado de edad para este subgrupo con la función weighted.mean()
weighted.mean(eph_ind$CH06[eph_ind$AGLOMERADO==18 & !is.na(eph_ind$IPCF) & eph_ind$CH04==2], na.rm = TRUE)## [1] 34Hicimos un calculo muy simple y ya vemos cómo el código se pone engorroso...
Qué pasaría si quisiéramos agregar mas condiciones, o si necesitamos realizar un calculo por grupo (por hogares por ejemplo) o si a continuación sobre este mismo grupo queremos aplicar otra operación
Manipular datos
Ahora calculemos el promedio ponderado de edad para este subgrupo con la función weighted.mean()
weighted.mean(eph_ind$CH06[eph_ind$AGLOMERADO==18 & !is.na(eph_ind$IPCF) & eph_ind$CH04==2], na.rm = TRUE)## [1] 34Hicimos un calculo muy simple y ya vemos cómo el código se pone engorroso...
Qué pasaría si quisiéramos agregar mas condiciones, o si necesitamos realizar un calculo por grupo (por hogares por ejemplo) o si a continuación sobre este mismo grupo queremos aplicar otra operación
Acá es donde entran las funciones, para facilitarnos las cosas
Manipular datos
Para manipular los datos empleamos la familia de funciones Tidyverse, que vendría a ser como una colección de paquetes que tienen en común que nos permiten realizar de manera eficiente un un amplio rango de operaciones.
Manipular datos
Para manipular los datos empleamos la familia de funciones Tidyverse, que vendría a ser como una colección de paquetes que tienen en común que nos permiten realizar de manera eficiente un un amplio rango de operaciones.
Entre los paquetes principales que usaremos están:
dplry: funciones con comandos claros para manipular basestidyr: limpieza y orden de los datosstringr: trabajar sobre strings con múltiples operaciones posiblesggplot2: graficar
Manipular datos
Como todo paquete para utilizarlo primero hay que instalarlo y luego cargarlo:
#install.packages("tidyverse")library(tidyverse)Manipular datos
Como todo paquete para utilizarlo primero hay que instalarlo y luego cargarlo:
#install.packages("tidyverse")library(tidyverse)Listo! Veamos ahora ya tenemos disponibles muchas de las principales funciones para trabajar con datos
Manipular datos
Como todo paquete para utilizarlo primero hay que instalarlo y luego cargarlo:
#install.packages("tidyverse")library(tidyverse)Listo! Veamos ahora ya tenemos disponibles muchas de las principales funciones para trabajar con datos
Solo nos falta una cosa, conocer cómo se usa y para qué sirve el elemento mas popular de esta librería...
Manipular datos
Como todo paquete para utilizarlo primero hay que instalarlo y luego cargarlo:
#install.packages("tidyverse")library(tidyverse)Listo! Veamos ahora ya tenemos disponibles muchas de las principales funciones para trabajar con datos
Solo nos falta una cosa, conocer cómo se usa y para qué sirve el elemento mas popular de esta librería... el operador "pipe" %>%
Operador pipe
El pipe es fundamental en R.
Operador pipe
El pipe es fundamental en R.
¿Por qué?
Operador pipe
El pipe es fundamental en R.
¿Por qué?
Porque permite unir acciones, es decir aplicar diversas funciones de forma secuencial sin tener que llamar siempre al dataframe
Operador pipe
El pipe es fundamental en R.
¿Por qué?
Porque permite unir acciones, es decir aplicar diversas funciones de forma secuencial sin tener que llamar siempre al dataframe
El operador pipe tiene la siguiente lógica:
Operador pipe
El pipe es fundamental en R.
¿Por qué?
Porque permite unir acciones, es decir aplicar diversas funciones de forma secuencial sin tener que llamar siempre al dataframe
El operador pipe tiene la siguiente lógica:
- Toma lo que esté a su izquierda
Operador pipe
El pipe es fundamental en R.
¿Por qué?
Porque permite unir acciones, es decir aplicar diversas funciones de forma secuencial sin tener que llamar siempre al dataframe
El operador pipe tiene la siguiente lógica:
- Toma lo que esté a su izquierda
- A ese objeto le aplica la función a su derecha
Operador pipe
El pipe es fundamental en R.
¿Por qué?
Porque permite unir acciones, es decir aplicar diversas funciones de forma secuencial sin tener que llamar siempre al dataframe
El operador pipe tiene la siguiente lógica:
- Toma lo que esté a su izquierda
- A ese objeto le aplica la función a su derecha
En la función ya está implícito el df sobre el que queremos realizar las transformaciones
Operador pipe
El pipe es fundamental en R.
¿Por qué?
Porque permite unir acciones, es decir aplicar diversas funciones de forma secuencial sin tener que llamar siempre al dataframe
El operador pipe tiene la siguiente lógica:
- Toma lo que esté a su izquierda
- A ese objeto le aplica la función a su derecha
En la función ya está implícito el df sobre el que queremos realizar las transformaciones
Veamos cómo funciona el pipe cuando empezamos a usar las funciones básicas y esenciales para trabajar con datos
EL ABC de dplyr
En esta clase veremos las funciones básicas y fundamentales del paquete dplyr que compone la familia de Tidyverse. Estas funciones son como el ABC para el procesamiento de datos
filter()select()rename()mutate()summarise()group_by()distinct()arrange()
EL ABC de dplyr
¿Qué quiero hacer? --> ¿Qué funcion necesito?
EL ABC de dplyr
¿Qué quiero hacer? --> ¿Qué funcion necesito?
| Función | Tarea |
|---|---|
filter() |
selecciona filas en base a una condición |
select() |
selecciona columnas (las mantiene o elimina) y también las ordena |
mutate() |
crea columnas nuevas y/o modifica las existentes |
rename() |
renombra las columnas |
summarise() |
convierte al dataframe una tabla resumen en base a distintos cálculos o estimaciones |
group_by() |
agrupa los datos según los valores que puede tomar una columna |
EL ABC de dplyr
Veamos paso a paso cada una, y luego veamos cómo concatenar cada una mediante el pipe
EL ABC de dplyr
Veamos paso a paso cada una, y luego veamos cómo concatenar cada una mediante el pipe
EL ABC de dplyr
Veamos paso a paso cada una, y luego veamos cómo concatenar cada una mediante el pipe
filter()select()rename()mutate()summarise()group_by()distinct()arrange()
Filter()
Usemos la opción filter() que como su nombre lo indica permite filtrar las filas de los datos en base a una condición
Esta función me pide dos argumentos básicos:
- dataframe a transformar
- condición a aplicar
rcia <- filter(eph_ind, AGLOMERADO==18)Estamos filtrando filas, quedándonos con observaciones que pertenezcan del aglomerado 18 (Resistencia - Chaco), y guardando ese subconjunto con un nuevo dataframe llamado "rcia"
Para estar seguros de que aplicamos correctamente el filtro podemos generar un tabulado de esa columna con table() y ver si hay valores distintos del buscado
table(rcia$AGLOMERADO)## ## 18 ## 1640Para estar seguros de que aplicamos correctamente el filtro podemos generar un tabulado de esa columna con table() y ver si hay valores distintos del buscado
table(rcia$AGLOMERADO)## ## 18 ## 1640Debería coincidir con el numero total de filas
nrow(rcia)## [1] 1640Para estar seguros de que aplicamos correctamente el filtro podemos generar un tabulado de esa columna con table() y ver si hay valores distintos del buscado
table(rcia$AGLOMERADO)## ## 18 ## 1640Debería coincidir con el numero total de filas
nrow(rcia)## [1] 1640Aplicamos correctamente nuestro filtro!
Filter()
Ahora usemos el pipe:
Filter()
Ahora usemos el pipe:
rcia <- eph_ind %>% filter(AGLOMERADO==18)Filter()
Ahora usemos el pipe:
rcia <- eph_ind %>% filter(AGLOMERADO==18)El pipe %>% toma el dataframe a la izquierda y le aplica la función a la derecha
Filter()
Ahora usemos el pipe:
rcia <- eph_ind %>% filter(AGLOMERADO==18)El pipe %>% toma el dataframe a la izquierda y le aplica la función a la derecha
Chequeemos que obtenemos el mismo resultado
table(rcia$AGLOMERADO)## ## 18 ## 1640nrow(rcia)## [1] 1640Filter()
Por supuesto que podemos pasarle mas condiciones al filtro
Retomemos nuestro ejemplo anterior, dejando solo los individuos del aglomerado 18 y que además que no tengan ingreso missing y que sean mujeres
Filter()
Por supuesto que podemos pasarle mas condiciones al filtro
Retomemos nuestro ejemplo anterior, dejando solo los individuos del aglomerado 18 y que además que no tengan ingreso missing y que sean mujeres
rcia <- eph_ind %>% filter(AGLOMERADO==18 & !is.na(IPCF) & CH04==2)Filter()
Por supuesto que podemos pasarle mas condiciones al filtro
Retomemos nuestro ejemplo anterior, dejando solo los individuos del aglomerado 18 y que además que no tengan ingreso missing y que sean mujeres
rcia <- eph_ind %>% filter(AGLOMERADO==18 & !is.na(IPCF) & CH04==2)Notar la diferencia con
rcia <- eph_ind[eph_ind$AGLOMERADO==18 & !is.na(eph_ind$IPCF) & eph_ind$CH04==2,]Filter()
Por supuesto que podemos pasarle mas condiciones al filtro
Retomemos nuestro ejemplo anterior, dejando solo los individuos del aglomerado 18 y que además que no tengan ingreso missing y que sean mujeres
rcia <- eph_ind %>% filter(AGLOMERADO==18 & !is.na(IPCF) & CH04==2)Notar la diferencia con
rcia <- eph_ind[eph_ind$AGLOMERADO==18 & !is.na(eph_ind$IPCF) & eph_ind$CH04==2,]Ya no es necesario llamar al dataframe antes llamar a la columna, el trabajo del pipe es ese, considerar el dataframe de la izquierda como implícito en la operación
Filter()
El símbolo & implica que ambas condiciones deben cumplirse. Así como este existen otros que nos permiten escribir las condiciones que necesitemos según el caso:
Filter()
El símbolo & implica que ambas condiciones deben cumplirse. Así como este existen otros que nos permiten escribir las condiciones que necesitemos según el caso:
| Símbolo | Condición |
|---|---|
| == | es igual a |
| != | es distinto a |
| < | es menor |
| <= | es menor igual |
| > | es menor |
| >= | es menor igual |
| %in% | se encuentra en o entre los valores |
| & | se cumpla una condición y la otra |
| | | se cumple una condición o la otra |
Filter()
Por ejemplo si a las condiciones anteriores quisiéramos agregar que sean personas jóvenes entre 18 y 30 años, escribiríamos
Filter()
Por ejemplo si a las condiciones anteriores quisiéramos agregar que sean personas jóvenes entre 18 y 30 años, escribiríamos
rcia <- eph_ind %>% filter(AGLOMERADO==18 & !is.na(IPCF) & CH04==2 & CH06 %in% (18:25))Pasemos a otra función
filter()select()rename()mutate()summarise()group_by()distinct()arrange()
Select()
Permite seleccionar las columnas de un dataframe y también ordenarlas
Select()
Permite seleccionar las columnas de un dataframe y también ordenarlas
La eph tiene muchas columnas, solo trabajaremos con algunas. Quedemosnos con las siguientes que usaremos
eph_short <- eph_ind %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, IPCF, CH04)Select()
Permite seleccionar las columnas de un dataframe y también ordenarlas
La eph tiene muchas columnas, solo trabajaremos con algunas. Quedemosnos con las siguientes que usaremos
eph_short <- eph_ind %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, IPCF, CH04)Ahora solo debemos tener estos nombres en la base
names(eph_short )## [1] "CODUSU" "NRO_HOGAR" "AGLOMERADO" "IPCF" "CH04"
Select()
Permite seleccionar las columnas de un dataframe y también ordenarlas
La eph tiene muchas columnas, solo trabajaremos con algunas. Quedemosnos con las siguientes que usaremos
eph_short <- eph_ind %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, IPCF, CH04)Ahora solo debemos tener estos nombres en la base
names(eph_short )## [1] "CODUSU" "NRO_HOGAR" "AGLOMERADO" "IPCF" "CH04"
Como hemos visto, también podemos aplicar la función sin el pipe
eph_short <- select(eph_ind, CODUSU, NRO_HOGAR, AGLOMERADO, IPCF, CH04)Pero...¿cómo haríamos si quisiéramos aplicar el filter() anterior junto con este select()?
Pero...¿cómo haríamos si quisiéramos aplicar el filter() anterior junto con este select()?
Armemos un data frame que:
- contenga las filas que cumplían con la condición anterior
- que tenga solo las columnas seleccionadas
Pero...¿cómo haríamos si quisiéramos aplicar el filter() anterior junto con este select()?
Armemos un data frame que:
- contenga las filas que cumplían con la condición anterior
- que tenga solo las columnas seleccionadas
rcia_short <- eph_ind %>% filter(AGLOMERADO==18 & !is.na(IPCF) & CH04==2) %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, IPCF, CH04)Pero...¿cómo haríamos si quisiéramos aplicar el filter() anterior junto con este select()?
Armemos un data frame que:
- contenga las filas que cumplían con la condición anterior
- que tenga solo las columnas seleccionadas
rcia_short <- eph_ind %>% filter(AGLOMERADO==18 & !is.na(IPCF) & CH04==2) %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, IPCF, CH04)El pipe me permite vincular operaciones generando un flujo donde el punto de partida y llegada es claro, y donde puedo leer fácilmente las operaciones aplicadas en el medio
Pero...¿cómo haríamos si quisiéramos aplicar el filter() anterior junto con este select()?
Armemos un data frame que:
- contenga las filas que cumplían con la condición anterior
- que tenga solo las columnas seleccionadas
rcia_short <- eph_ind %>% filter(AGLOMERADO==18 & !is.na(IPCF) & CH04==2) %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, IPCF, CH04)El pipe me permite vincular operaciones generando un flujo donde el punto de partida y llegada es claro, y donde puedo leer fácilmente las operaciones aplicadas en el medio
dim(rcia_short)## [1] 858 5Si al principio no resulta tan intuitivo entender las operaciones unidas por el pipe, podemos cortarlo en partes mas pequeñas e ir visualizando cada output hasta llegar al final, entendiendo cada paso intermedio
Si al principio no resulta tan intuitivo entender las operaciones unidas por el pipe, podemos cortarlo en partes mas pequeñas e ir visualizando cada output hasta llegar al final, entendiendo cada paso intermedio
Luego de varias veces iremos ganando gimnasia en esto.
Si al principio no resulta tan intuitivo entender las operaciones unidas por el pipe, podemos cortarlo en partes mas pequeñas e ir visualizando cada output hasta llegar al final, entendiendo cada paso intermedio
Luego de varias veces iremos ganando gimnasia en esto.
Para visualizar más fácilmente lo que ocurre con los datos en este caso, intercambiemos el orden seleccionando primero las columnas con select() y luego las filas mediante las condiciones del filter()
Si al principio no resulta tan intuitivo entender las operaciones unidas por el pipe, podemos cortarlo en partes mas pequeñas e ir visualizando cada output hasta llegar al final, entendiendo cada paso intermedio
Luego de varias veces iremos ganando gimnasia en esto.
Para visualizar más fácilmente lo que ocurre con los datos en este caso, intercambiemos el orden seleccionando primero las columnas con select() y luego las filas mediante las condiciones del filter()
Visualicemos cada paso del código
rcia_short <- eph_ind %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, IPCF, CH04) %>% filter(AGLOMERADO==18 & !is.na(IPCF) & CH04==2)Empezamos llamando al data frame eph_ind que tiene todas las filas y columnas
rcia_short <- eph_ind| CODUSU | ANO4 | TRIMESTRE | NRO_HOGAR | COMPONENTE | H15 | REGION | MAS_500 | AGLOMERADO | PONDERA | CH03 | CH04 | CH05 | CH06 | CH07 |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| TQRMNORVQHJNLTCDEGGFB00788741 | 2022 | 3 | 1 | 1 | 1 | 42 | S | 10 | 537 | 1 | 1 | 10/04/1961 | 61 | 5 |
| TQRMNOQSWHKNKRCDEGJBF00789123 | 2022 | 3 | 1 | 1 | 1 | 43 | S | 13 | 543 | 1 | 1 | 06/10/1953 | 68 | 2 |
| TQRMNOQSWHKNKRCDEGJBF00789123 | 2022 | 3 | 1 | 2 | 1 | 43 | S | 13 | 543 | 2 | 2 | 28/08/1955 | 66 | 2 |
| TQRMNOTSQHLLMLCDEIIAD00703213 | 2022 | 3 | 1 | 1 | 1 | 1 | S | 32 | 894 | 1 | 1 | 10/03/1957 | 65 | 2 |
| TQRMNOTSQHLLMLCDEIIAD00703213 | 2022 | 3 | 1 | 2 | 1 | 1 | S | 32 | 894 | 2 | 2 | 07/01/1958 | 64 | 2 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 2022 | 3 | 1 | 1 | 1 | 40 | S | 29 | 345 | 1 | 2 | 18/09/1977 | 44 | 5 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 2022 | 3 | 1 | 2 | 1 | 40 | S | 29 | 345 | 3 | 1 | 13/05/2003 | 19 | 1 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 2022 | 3 | 1 | 3 | 1 | 40 | S | 29 | 345 | 3 | 1 | 30/03/2010 | 12 | 5 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 2022 | 3 | 1 | 4 | 0 | 40 | S | 29 | 345 | 3 | 1 | 06/03/2015 | 7 | 5 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 2022 | 3 | 1 | 5 | 1 | 40 | S | 29 | 345 | 4 | 2 | 30/04/2005 | 17 | 1 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 2022 | 3 | 1 | 1 | 1 | 44 | N | 91 | 141 | 1 | 1 | 17/08/1970 | 52 | 2 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 2022 | 3 | 1 | 2 | 1 | 44 | N | 91 | 141 | 2 | 2 | 03/07/1974 | 48 | 2 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 2022 | 3 | 1 | 3 | 1 | 44 | N | 91 | 141 | 3 | 1 | 16/08/2007 | 15 | 5 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 2022 | 3 | 1 | 4 | 1 | 44 | N | 91 | 141 | 3 | 1 | 17/09/2010 | 11 | 5 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 2022 | 3 | 1 | 1 | 1 | 41 | N | 7 | 303 | 1 | 1 | 19/08/1968 | 54 | 1 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 2022 | 3 | 1 | 2 | 1 | 41 | N | 7 | 303 | 2 | 2 | 28/05/1970 | 52 | 1 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 2022 | 3 | 1 | 3 | 1 | 41 | N | 7 | 303 | 3 | 2 | 09/01/1997 | 25 | 5 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 2022 | 3 | 1 | 4 | 1 | 41 | N | 7 | 303 | 3 | 2 | 08/11/2003 | 18 | 5 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 2022 | 3 | 1 | 5 | 0 | 41 | N | 7 | 303 | 5 | 1 | 12/05/2014 | 8 | 5 |
| TQRMNOPXUHLMKUCDEGOIH00706727 | 2022 | 3 | 1 | 1 | 1 | 40 | N | 18 | 275 | 1 | 1 | 03/03/1983 | 39 | 2 |
| TQRMNOPXUHLMKUCDEGOIH00706727 | 2022 | 3 | 1 | 2 | 1 | 40 | N | 18 | 275 | 2 | 2 | 26/08/1985 | 37 | 2 |
| TQRMNOPXUHLMKUCDEGOIH00706727 | 2022 | 3 | 1 | 3 | 1 | 40 | N | 18 | 275 | 3 | 1 | 04/02/2012 | 10 | 5 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 2022 | 3 | 1 | 1 | 1 | 41 | N | 12 | 333 | 1 | 1 | 20/12/1994 | 27 | 1 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 2022 | 3 | 1 | 2 | 1 | 41 | N | 12 | 333 | 2 | 2 | 29/11/1993 | 28 | 1 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 2022 | 3 | 1 | 3 | 0 | 41 | N | 12 | 333 | 3 | 2 | 28/11/2014 | 7 | 5 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 2022 | 3 | 1 | 4 | 0 | 41 | N | 12 | 333 | 3 | 2 | 29/08/2012 | 9 | 5 |
| TQRMNOQSUHMLKTCDEHIBB00717071 | 2022 | 3 | 1 | 1 | 1 | 40 | N | 22 | 108 | 1 | 2 | 01/05/1988 | 34 | 1 |
| TQRMNOQSUHMLKTCDEHIBB00717071 | 2022 | 3 | 1 | 2 | 1 | 40 | N | 22 | 108 | 2 | 1 | 19/02/1992 | 30 | 1 |
| TQRMNOQSQHMLKTCDEHIBB00709425 | 2022 | 3 | 1 | 1 | 1 | 40 | N | 22 | 143 | 1 | 2 | 01/11/1976 | 45 | 5 |
| TQRMNOQSQHMLKTCDEHIBB00709425 | 2022 | 3 | 1 | 2 | 1 | 40 | N | 22 | 143 | 3 | 1 | 27/10/1997 | 24 | 5 |
| TQRMNOQSQHMLKTCDEHIBB00709425 | 2022 | 3 | 1 | 3 | 1 | 40 | N | 22 | 143 | 3 | 1 | 27/10/1997 | 24 | 5 |
| TQRMNOQSQHMLKTCDEHIBB00709425 | 2022 | 3 | 1 | 4 | 1 | 40 | N | 22 | 143 | 3 | 2 | 11/08/1999 | 23 | 5 |
| TQRMNOQTVHJMKTCDEHIBB00784254 | 2022 | 3 | 1 | 1 | 1 | 40 | N | 22 | 118 | 1 | 2 | 21/09/1951 | 70 | 3 |
| TQRMNOQTVHJMKTCDEHIBB00784254 | 2022 | 3 | 1 | 2 | 1 | 40 | N | 22 | 118 | 3 | 1 | 05/09/1986 | 36 | 5 |
| TQRMNOQXUHLMRQCDEIJAH00706181 | 2022 | 3 | 1 | 1 | 1 | 1 | S | 33 | 1792 | 1 | 1 | 09/12/1962 | 59 | 1 |
| TQRMNOQXUHLMRQCDEIJAH00706181 | 2022 | 3 | 1 | 2 | 1 | 1 | S | 33 | 1792 | 2 | 2 | 13/04/1960 | 62 | 1 |
| TQRMNORPQHLMKSCDEHJGH00790306 | 2022 | 3 | 1 | 1 | 1 | 40 | S | 23 | 220 | 1 | 1 | 03/04/1950 | 72 | 2 |
| TQRMNORPQHLMKSCDEHJGH00790306 | 2022 | 3 | 1 | 2 | 1 | 40 | S | 23 | 220 | 2 | 2 | 26/03/1955 | 67 | 2 |
| TQRMNORPQHLMKSCDEHJGH00790306 | 2022 | 3 | 1 | 3 | 1 | 40 | S | 23 | 220 | 3 | 2 | 23/09/1983 | 38 | 5 |
| TQRMNORPQHLMKSCDEHJGH00790306 | 2022 | 3 | 1 | 4 | 1 | 40 | S | 23 | 220 | 3 | 2 | 05/06/1982 | 40 | 5 |
| TQRMNORPQHLMKSCDEHJGH00790306 | 2022 | 3 | 1 | 5 | 1 | 40 | S | 23 | 220 | 3 | 2 | 08/11/1987 | 34 | 5 |
| TQRMNORUPHJMKUCDEHGHJ00784183 | 2022 | 3 | 1 | 1 | 1 | 44 | N | 20 | 134 | 1 | 1 | 01/01/1900 | 73 | 2 |
| TQRMNORUPHJMKUCDEHGHJ00784183 | 2022 | 3 | 1 | 2 | 1 | 44 | N | 20 | 134 | 2 | 2 | 01/01/1900 | 76 | 2 |
| TQRMNOQXUHMLSNCDEIJAH00719506 | 2022 | 3 | 1 | 1 | 1 | 1 | S | 33 | 5562 | 1 | 2 | 03/07/1941 | 81 | 4 |
| TQRMNOQXUHMLSNCDEIJAH00719506 | 2022 | 3 | 1 | 2 | 1 | 1 | S | 33 | 5562 | 3 | 1 | 15/08/1966 | 56 | 5 |
| TQRMNOTXVHJOPSCDEIJAH00786237 | 2022 | 3 | 2 | 1 | 1 | 1 | S | 33 | 2433 | 1 | 1 | 01/07/1949 | 73 | 1 |
| TQRMNOTXVHJOPSCDEIJAH00786237 | 2022 | 3 | 2 | 2 | 1 | 1 | S | 33 | 2433 | 2 | 2 | 01/03/1953 | 69 | 1 |
| TQRMNOQYPHMOKRCDEGLDF00713344 | 2022 | 3 | 1 | 1 | 1 | 41 | N | 15 | 93 | 1 | 2 | 18/04/1953 | 69 | 4 |
| TQRMNOQYPHMOKRCDEGLDF00713344 | 2022 | 3 | 1 | 2 | 1 | 41 | N | 15 | 93 | 3 | 2 | 03/10/1982 | 39 | 5 |
| TQRMNORPUHJNOLCDEIIAD00785503 | 2022 | 3 | 1 | 1 | 1 | 1 | S | 32 | 2030 | 1 | 1 | 15/03/1991 | 31 | 5 |
(aquí imprimiremos a modo ilustrativo solo las primeras 50 filas y 15 columnas)
Luego nos quedamos solo con algunas columnas, pero misma cantidad de filas
rcia_short <- eph_ind %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, IPCF, CH04)| CODUSU | NRO_HOGAR | AGLOMERADO | IPCF | CH04 |
|---|---|---|---|---|
| TQRMNORVQHJNLTCDEGGFB00788741 | 1 | 10 | 46000.00 | 1 |
| TQRMNOQSWHKNKRCDEGJBF00789123 | 1 | 13 | 160200.00 | 1 |
| TQRMNOQSWHKNKRCDEGJBF00789123 | 1 | 13 | 160200.00 | 2 |
| TQRMNOTSQHLLMLCDEIIAD00703213 | 1 | 32 | 0.00 | 1 |
| TQRMNOTSQHLLMLCDEIIAD00703213 | 1 | 32 | 0.00 | 2 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 1 | 29 | 10360.00 | 2 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 1 | 29 | 10360.00 | 1 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 1 | 29 | 10360.00 | 1 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 1 | 29 | 10360.00 | 1 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 1 | 29 | 10360.00 | 2 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 1 | 91 | 42000.00 | 1 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 1 | 91 | 42000.00 | 2 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 1 | 91 | 42000.00 | 1 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 1 | 91 | 42000.00 | 1 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 1 | 7 | 23020.00 | 1 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 1 | 7 | 23020.00 | 2 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 1 | 7 | 23020.00 | 2 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 1 | 7 | 23020.00 | 2 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 1 | 7 | 23020.00 | 1 |
| TQRMNOPXUHLMKUCDEGOIH00706727 | 1 | 18 | 26666.67 | 1 |
| TQRMNOPXUHLMKUCDEGOIH00706727 | 1 | 18 | 26666.67 | 2 |
| TQRMNOPXUHLMKUCDEGOIH00706727 | 1 | 18 | 26666.67 | 1 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 1 | 12 | 37950.00 | 1 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 1 | 12 | 37950.00 | 2 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 1 | 12 | 37950.00 | 2 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 1 | 12 | 37950.00 | 2 |
| TQRMNOQSUHMLKTCDEHIBB00717071 | 1 | 22 | 97500.00 | 2 |
| TQRMNOQSUHMLKTCDEHIBB00717071 | 1 | 22 | 97500.00 | 1 |
| TQRMNOQSQHMLKTCDEHIBB00709425 | 1 | 22 | 31000.00 | 2 |
| TQRMNOQSQHMLKTCDEHIBB00709425 | 1 | 22 | 31000.00 | 1 |
| TQRMNOQSQHMLKTCDEHIBB00709425 | 1 | 22 | 31000.00 | 1 |
| TQRMNOQSQHMLKTCDEHIBB00709425 | 1 | 22 | 31000.00 | 2 |
| TQRMNOQTVHJMKTCDEHIBB00784254 | 1 | 22 | 62500.00 | 2 |
| TQRMNOQTVHJMKTCDEHIBB00784254 | 1 | 22 | 62500.00 | 1 |
| TQRMNOQXUHLMRQCDEIJAH00706181 | 1 | 33 | 39500.00 | 1 |
| TQRMNOQXUHLMRQCDEIJAH00706181 | 1 | 33 | 39500.00 | 2 |
| TQRMNORPQHLMKSCDEHJGH00790306 | 1 | 23 | 0.00 | 1 |
| TQRMNORPQHLMKSCDEHJGH00790306 | 1 | 23 | 0.00 | 2 |
| TQRMNORPQHLMKSCDEHJGH00790306 | 1 | 23 | 0.00 | 2 |
| TQRMNORPQHLMKSCDEHJGH00790306 | 1 | 23 | 0.00 | 2 |
| TQRMNORPQHLMKSCDEHJGH00790306 | 1 | 23 | 0.00 | 2 |
| TQRMNORUPHJMKUCDEHGHJ00784183 | 1 | 20 | 81000.00 | 1 |
| TQRMNORUPHJMKUCDEHGHJ00784183 | 1 | 20 | 81000.00 | 2 |
| TQRMNOQXUHMLSNCDEIJAH00719506 | 1 | 33 | 62500.00 | 2 |
| TQRMNOQXUHMLSNCDEIJAH00719506 | 1 | 33 | 62500.00 | 1 |
| TQRMNOTXVHJOPSCDEIJAH00786237 | 2 | 33 | 32000.00 | 1 |
| TQRMNOTXVHJOPSCDEIJAH00786237 | 2 | 33 | 32000.00 | 2 |
| TQRMNOQYPHMOKRCDEGLDF00713344 | 1 | 15 | 27750.00 | 2 |
| TQRMNOQYPHMOKRCDEGLDF00713344 | 1 | 15 | 27750.00 | 2 |
| TQRMNORPUHJNOLCDEIIAD00785503 | 1 | 32 | 110000.00 | 1 |
Finalmente nos quedamos con las filas que cumplen las condiciones que impusimos
rcia_short <- eph_ind %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, IPCF, CH04) %>% filter(AGLOMERADO==18 & !is.na(IPCF) & CH04==2)| CODUSU | NRO_HOGAR | AGLOMERADO | IPCF | CH04 |
|---|---|---|---|---|
| TQRMNOPXUHLMKUCDEGOIH00706727 | 1 | 18 | 26666.67 | 2 |
| TQRMNOQQWHKOKRCDEGOIH00789766 | 1 | 18 | 20500.00 | 2 |
| TQRMNOQUQHLOKSCDEGOIH00706615 | 1 | 18 | 31540.00 | 2 |
| TQRMNOQUQHLOKSCDEGOIH00706615 | 1 | 18 | 31540.00 | 2 |
| TQRMNOQUQHLOKSCDEGOIH00706615 | 1 | 18 | 31540.00 | 2 |
| TQRMNOQUTHMMKUCDEGOIH00714113 | 1 | 18 | 39357.14 | 2 |
| TQRMNOQUTHMMKUCDEGOIH00714113 | 1 | 18 | 39357.14 | 2 |
| TQRMNOQUTHMMKUCDEGOIH00714113 | 1 | 18 | 39357.14 | 2 |
| TQRMNOQTYHLOKSCDEGOIH00707817 | 1 | 18 | 11300.00 | 2 |
| TQRMNOQTYHLOKSCDEGOIH00707817 | 1 | 18 | 11300.00 | 2 |
| TQRMNOQTYHLOKSCDEGOIH00707817 | 1 | 18 | 11300.00 | 2 |
| TQRMNORUXHJOKMCDEGOIH00783893 | 1 | 18 | 11666.67 | 2 |
| TQRMNORUXHJOKMCDEGOIH00783893 | 1 | 18 | 11666.67 | 2 |
| TQRMNORUXHJOKMCDEGOIH00783893 | 1 | 18 | 11666.67 | 2 |
| TQRMNOQWRHMMLOCDEGOIH00710799 | 1 | 18 | 29083.33 | 2 |
| TQRMNOQWRHMMLOCDEGOIH00710799 | 1 | 18 | 29083.33 | 2 |
| TQRMNOQWRHMMLOCDEGOIH00710799 | 1 | 18 | 29083.33 | 2 |
| TQRMNOQWRHMMLOCDEGOIH00710799 | 1 | 18 | 29083.33 | 2 |
| TQRMNOSUUHKOKQCDEGOIH00789706 | 2 | 18 | 8850.00 | 2 |
| TQRMNOSUUHKOKQCDEGOIH00789706 | 2 | 18 | 8850.00 | 2 |
| TQRMNOSUUHKOKQCDEGOIH00789706 | 2 | 18 | 8850.00 | 2 |
| TQRMNOPWYHLOKMCDEGOIH00707843 | 1 | 18 | 31583.33 | 2 |
| TQRMNOPWYHLOKMCDEGOIH00707843 | 1 | 18 | 31583.33 | 2 |
| TQRMNOPWYHLOKMCDEGOIH00707843 | 1 | 18 | 31583.33 | 2 |
| TQRMNOQRVHLOLOCDEGOIH00706471 | 1 | 18 | 35900.00 | 2 |
| TQRMNOQRVHLOLOCDEGOIH00706471 | 1 | 18 | 35900.00 | 2 |
| TQRMNOQRVHLOLOCDEGOIH00706471 | 1 | 18 | 35900.00 | 2 |
| TQRMNOQRVHLOLOCDEGOIH00706471 | 1 | 18 | 35900.00 | 2 |
| TQRMNOPVQHLMLPCDEGOIH00706831 | 1 | 18 | 45000.00 | 2 |
| TQRMNORPQHMMLMCDEGOIH00711885 | 1 | 18 | 64700.00 | 2 |
| TQRMNORPYHMOLPCDEGOIH00714204 | 1 | 18 | 35000.00 | 2 |
| TQRMNOQRXHLOLOCDEGOIH00706675 | 1 | 18 | 14750.00 | 2 |
| TQRMNOSUUHLOKSCDEGOIH00783865 | 1 | 18 | 110000.00 | 2 |
| TQRMNOQPPHMOLPCDEGOIH00713798 | 1 | 18 | 25000.00 | 2 |
| TQRMNOQPPHMOLPCDEGOIH00713798 | 1 | 18 | 25000.00 | 2 |
| TQRMNOQXUHKLLQCDEGOIH00789760 | 1 | 18 | 0.00 | 2 |
| TQRMNOPXVHLLLQCDEGOIH00705672 | 1 | 18 | 30000.00 | 2 |
| TQRMNOQRWHLOLOCDEGOIH00706647 | 1 | 18 | 24250.00 | 2 |
| TQRMNOSRYHLLLQCDEGOIH00705677 | 1 | 18 | 23333.33 | 2 |
| TQRMNOSRYHLLLQCDEGOIH00705677 | 1 | 18 | 23333.33 | 2 |
| TQRMNOVSPHKOKPCDEGOIH00789764 | 1 | 18 | 19258.33 | 2 |
| TQRMNOVSPHKOKPCDEGOIH00789764 | 1 | 18 | 19258.33 | 2 |
| TQRMNOVSPHKOKPCDEGOIH00789764 | 1 | 18 | 19258.33 | 2 |
| TQRMNOVSPHKOKPCDEGOIH00789764 | 1 | 18 | 19258.33 | 2 |
| TQRMNOVRYHKOKPCDEGOIH00789765 | 1 | 18 | 9400.00 | 2 |
| TQRMNORWTHJMKOCDEGOIH00783880 | 1 | 18 | 100833.33 | 2 |
| TQRMNOSUSHLOKSCDEGOIH00711215 | 1 | 18 | 30140.00 | 2 |
| TQRMNOSUSHLOKSCDEGOIH00711215 | 1 | 18 | 30140.00 | 2 |
| TQRMNOSUSHLOKSCDEGOIH00711215 | 1 | 18 | 30140.00 | 2 |
| TQRMNOQTXHLOKSCDEGOIH00706614 | 1 | 18 | 27200.00 | 2 |
Select()
Esta misma función también me permite reordenar las filas
Select()
Esta misma función también me permite reordenar las filas
Supongamos que quiero traer adelante las filas sobre las que voy a aplicar una operación y dejar atrás los id
eph_short <- eph_ind %>% select(AGLOMERADO, IPCF, CH04, CODUSU, NRO_HOGAR)Select()
Esta misma función también me permite reordenar las filas
Supongamos que quiero traer adelante las filas sobre las que voy a aplicar una operación y dejar atrás los id
eph_short <- eph_ind %>% select(AGLOMERADO, IPCF, CH04, CODUSU, NRO_HOGAR)names(eph_short)## [1] "AGLOMERADO" "IPCF" "CH04" "CODUSU" "NRO_HOGAR"Un atajo para no especificar todas las demás columnas es es la función everything() dentro del select()
eph_short <- eph_ind %>% select(AGLOMERADO, IPCF, CH04, everything())Un atajo para no especificar todas las demás columnas es es la función everything() dentro del select()
eph_short <- eph_ind %>% select(AGLOMERADO, IPCF, CH04, everything())names(eph_short)## [1] "AGLOMERADO" "IPCF" "CH04" "CODUSU" "ANO4" ## [6] "TRIMESTRE" "NRO_HOGAR" "COMPONENTE" "H15" "REGION" ## [11] "MAS_500" "PONDERA" "CH03" "CH05" "CH06" ## [16] "CH07" "CH08" "CH09" "CH10" "CH11" ## [21] "CH12" "CH13" "CH14" "CH15" "CH15_COD" ## [26] "CH16" "CH16_COD" "NIVEL_ED" "ESTADO" "CAT_OCUP" ## [31] "CAT_INAC" "IMPUTA" "PP02C1" "PP02C2" "PP02C3" ## [36] "PP02C4" "PP02C5" "PP02C6" "PP02C7" "PP02C8" ## [41] "PP02E" "PP02H" "PP02I" "PP03C" "PP03D" ## [46] "PP3E_TOT" "PP3F_TOT" "PP03G" "PP03H" "PP03I" ## [51] "PP03J" "INTENSI" "PP04A" "PP04B_COD" "PP04B1" ## [56] "PP04B2" "PP04B3_MES" "PP04B3_ANO" "PP04B3_DIA" "PP04C" ## [61] "PP04C99" "PP04D_COD" "PP04G" "PP05B2_MES" "PP05B2_ANO"## [66] "PP05B2_DIA" "PP05C_1" "PP05C_2" "PP05C_3" "PP05E" ## [71] "PP05F" "PP05H" "PP06A" "PP06C" "PP06D" ## [76] "PP06E" "PP06H" "PP07A" "PP07C" "PP07D" ## [81] "PP07E" "PP07F1" "PP07F2" "PP07F3" "PP07F4" ## [86] "PP07F5" "PP07G1" "PP07G2" "PP07G3" "PP07G4" ## [91] "PP07G_59" "PP07H" "PP07I" "PP07J" "PP07K" ## [96] "PP08D1" "PP08D4" "PP08F1" "PP08F2" "PP08J1" ## [101] "PP08J2" "PP08J3" "PP09A" "PP09A_ESP" "PP09B" ## [106] "PP09C" "PP09C_ESP" "PP10A" "PP10C" "PP10D" ## [111] "PP10E" "PP11A" "PP11B_COD" "PP11B1" "PP11B2_MES"## [116] "PP11B2_ANO" "PP11B2_DIA" "PP11C" "PP11C99" "PP11D_COD" ## [121] "PP11G_ANO" "PP11G_MES" "PP11G_DIA" "PP11L" "PP11L1" ## [126] "PP11M" "PP11N" "PP11O" "PP11P" "PP11Q" ## [131] "PP11R" "PP11S" "PP11T" "P21" "DECOCUR" ## [136] "IDECOCUR" "RDECOCUR" "GDECOCUR" "PDECOCUR" "ADECOCUR" ## [141] "PONDIIO" "TOT_P12" "P47T" "DECINDR" "IDECINDR" ## [146] "RDECINDR" "GDECINDR" "PDECINDR" "ADECINDR" "PONDII" ## [151] "V2_M" "V3_M" "V4_M" "V5_M" "V8_M" ## [156] "V9_M" "V10_M" "V11_M" "V12_M" "V18_M" ## [161] "V19_AM" "V21_M" "T_VI" "ITF" "DECIFR" ## [166] "IDECIFR" "RDECIFR" "GDECIFR" "PDECIFR" "ADECIFR" ## [171] "DECCFR" "IDECCFR" "RDECCFR" "GDECCFR" "PDECCFR" ## [176] "ADECCFR" "PONDIH"También es posible usar los : para seleccionar un rango de columnas consecutivas
eph_short <- eph_ind %>% select(AGLOMERADO, IPCF, CH04, NIVEL_ED:CAT_INAC)También es posible usar los : para seleccionar un rango de columnas consecutivas
eph_short <- eph_ind %>% select(AGLOMERADO, IPCF, CH04, NIVEL_ED:CAT_INAC)names(eph_short)## [1] "AGLOMERADO" "IPCF" "CH04" "NIVEL_ED" "ESTADO" ## [6] "CAT_OCUP" "CAT_INAC"
También es posible usar los : para seleccionar un rango de columnas consecutivas
eph_short <- eph_ind %>% select(AGLOMERADO, IPCF, CH04, NIVEL_ED:CAT_INAC)names(eph_short)## [1] "AGLOMERADO" "IPCF" "CH04" "NIVEL_ED" "ESTADO" ## [6] "CAT_OCUP" "CAT_INAC"
Incluso podemos hacer esto mismo indicando las columnas con subindices de números y usando :
eph_short <- eph_ind %>% select(9, 170, 12, 27:31)También es posible usar los : para seleccionar un rango de columnas consecutivas
eph_short <- eph_ind %>% select(AGLOMERADO, IPCF, CH04, NIVEL_ED:CAT_INAC)names(eph_short)## [1] "AGLOMERADO" "IPCF" "CH04" "NIVEL_ED" "ESTADO" ## [6] "CAT_OCUP" "CAT_INAC"
Incluso podemos hacer esto mismo indicando las columnas con subindices de números y usando :
eph_short <- eph_ind %>% select(9, 170, 12, 27:31)names(eph_short)## [1] "AGLOMERADO" "IPCF" "CH04" "NIVEL_ED" "ESTADO" ## [6] "CAT_OCUP" "CAT_INAC" "IMPUTA"Select()
La misma función nos permite también borrar columnas, para ello solo anteponemos -
Select()
La misma función nos permite también borrar columnas, para ello solo anteponemos -
Para borrar por ejemplo la columna AGLOMERADO, sería simplemente
eph_short <- eph_short %>% select(-AGLOMERADO)Select()
La misma función nos permite también borrar columnas, para ello solo anteponemos -
Para borrar por ejemplo la columna AGLOMERADO, sería simplemente
eph_short <- eph_short %>% select(-AGLOMERADO)## [1] "IPCF" "CH04" "NIVEL_ED" "ESTADO" "CAT_OCUP" "CAT_INAC" "IMPUTA"De la misma manera que antes podemos borrar un conjunto de columnas con : y ()
eph_short <- eph_short %>% select(-(NIVEL_ED:CAT_INAC), -IPCF)Select()
La misma función nos permite también borrar columnas, para ello solo anteponemos -
Para borrar por ejemplo la columna AGLOMERADO, sería simplemente
eph_short <- eph_short %>% select(-AGLOMERADO)## [1] "IPCF" "CH04" "NIVEL_ED" "ESTADO" "CAT_OCUP" "CAT_INAC" "IMPUTA"De la misma manera que antes podemos borrar un conjunto de columnas con : y ()
eph_short <- eph_short %>% select(-(NIVEL_ED:CAT_INAC), -IPCF)## [1] "CH04" "IMPUTA"(También es posible borrar usando los números de columnas)
Select()
Así como la función everything() existen otras que se combinan muy bien con select() para poder manipular los datos

Fuente:https://rsanchezs.gitbooks.io/rprogramming/content/chapter9/select.html
Select()
Veamos un ejemplo: en el diseño de registro del EPH todas las preguntas que empiezan con "PP08" refieren a la ocupación principal de los asalariados.
Select()
Veamos un ejemplo: en el diseño de registro del EPH todas las preguntas que empiezan con "PP08" refieren a la ocupación principal de los asalariados.
Si estamos interesados en mantener solo este set de preguntas en nuestra base podemos lograrlo fácilmente
Select()
Veamos un ejemplo: en el diseño de registro del EPH todas las preguntas que empiezan con "PP08" refieren a la ocupación principal de los asalariados.
Si estamos interesados en mantener solo este set de preguntas en nuestra base podemos lograrlo fácilmente
eph_asal <- eph_ind %>% select(CODUSU, NRO_HOGAR, starts_with("PP08"))Select()
Veamos un ejemplo: en el diseño de registro del EPH todas las preguntas que empiezan con "PP08" refieren a la ocupación principal de los asalariados.
Si estamos interesados en mantener solo este set de preguntas en nuestra base podemos lograrlo fácilmente
eph_asal <- eph_ind %>% select(CODUSU, NRO_HOGAR, starts_with("PP08"))names(eph_short)## [1] "CH04" "IMPUTA"filter()select()rename()mutate()summarise()group_by()distinct()arrange()
Rename()
Como su nombre lo indica permite renombrar las variables
Rename()
Como su nombre lo indica permite renombrar las variables
Rename()
Como su nombre lo indica permite renombrar las variables
rcia_short <- eph_ind %>% filter(AGLOMERADO==18 & !is.na(IPCF) & CH04==2) %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, IPCF, CH04, CH06) %>% rename(sexo = CH04, edad = CH06)Rename()
Como su nombre lo indica permite renombrar las variables
rcia_short <- eph_ind %>% filter(AGLOMERADO==18 & !is.na(IPCF) & CH04==2) %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, IPCF, CH04, CH06) %>% rename(sexo = CH04, edad = CH06)## CODUSU NRO_HOGAR AGLOMERADO IPCF sexo edad## 1 TQRMNOPXUHLMKUCDEGOIH00706727 1 18 26666.67 2 37## 2 TQRMNOQQWHKOKRCDEGOIH00789766 1 18 20500.00 2 26## 3 TQRMNOQUQHLOKSCDEGOIH00706615 1 18 31540.00 2 65## 4 TQRMNOQUQHLOKSCDEGOIH00706615 1 18 31540.00 2 32## 5 TQRMNOQUQHLOKSCDEGOIH00706615 1 18 31540.00 2 44Rename()
Nos quedaron columnas en mayúsculas y en minúsculas
Una extensión de esta función, rename_with() nos permite renombrar varias columnas a la vez
rcia_short <- eph_ind %>% filter(AGLOMERADO==18 & !is.na(IPCF) & CH04==2) %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, IPCF, CH04, CH06) %>% rename(sexo = CH04, edad = CH06) %>% rename_with(tolower)Rename()
Nos quedaron columnas en mayúsculas y en minúsculas
Una extensión de esta función, rename_with() nos permite renombrar varias columnas a la vez
rcia_short <- eph_ind %>% filter(AGLOMERADO==18 & !is.na(IPCF) & CH04==2) %>% select(CODUSU, NRO_HOGAR, AGLOMERADO, IPCF, CH04, CH06) %>% rename(sexo = CH04, edad = CH06) %>% rename_with(tolower)names(rcia_short)## [1] "codusu" "nro_hogar" "aglomerado" "ipcf" "sexo" ## [6] "edad"Rename()
Podemos realizar incluso operaciones un poco mas complejas. Para eso es importante conocer qué argumentos debemos especificar
rename_with(.data, .fn, .cols, ...)
Rename()
Podemos realizar incluso operaciones un poco mas complejas. Para eso es importante conocer qué argumentos debemos especificar
rename_with(.data, .fn, .cols, ...)
Esto se lee como:
- debo indicarle sobre qué dataframe operar:
.data - qué función aplicar:
.fn - sobre cuáles columnas:
.cols
Rename()
Podemos realizar incluso operaciones un poco mas complejas. Para eso es importante conocer qué argumentos debemos especificar
rename_with(.data, .fn, .cols, ...)
Esto se lee como:
- debo indicarle sobre qué dataframe operar:
.data - qué función aplicar:
.fn - sobre cuáles columnas:
.cols
Veamos un ejemplo, supongamos que antes de juntar la base de individuos con la de hogares, quiero identificar a las variables de cada base.
Rename()
Podemos realizar incluso operaciones un poco mas complejas. Para eso es importante conocer qué argumentos debemos especificar
rename_with(.data, .fn, .cols, ...)
Esto se lee como:
- debo indicarle sobre qué dataframe operar:
.data - qué función aplicar:
.fn - sobre cuáles columnas:
.cols
Veamos un ejemplo, supongamos que antes de juntar la base de individuos con la de hogares, quiero identificar a las variables de cada base.
Para ello buscamos agregar el sufijo _ind a todas las variables
Cómo haríamos?
Vamos por parte
.data: no debo especificar, de eso se encarga %>%.cols: quiero hacerlo sobre todas, entonces puedo usar la funcióneverything().fn: tengo que pensar una función que a lo que ya tengo le agregue un sufijo
Vamos por parte
.data: no debo especificar, de eso se encarga %>%.cols: quiero hacerlo sobre todas, entonces puedo usar la funcióneverything().fn: tengo que pensar una función que a lo que ya tengo le agregue un sufijo
Para esto último nos ayuda la función paste()
Vamos por parte
.data: no debo especificar, de eso se encarga %>%.cols: quiero hacerlo sobre todas, entonces puedo usar la funcióneverything().fn: tengo que pensar una función que a lo que ya tengo le agregue un sufijo
Para esto último nos ayuda la función paste()
eph_ind <- eph_ind %>% rename_with( .cols = everything(), .fn=function(x){ paste0(x, "_ind") } )Hacia el final del curso podremos entender mejor como opera y cuando necesitamos funciones del tipo function(x){ paste0(x, "_ind") }
Por ahora, elijan creer
names(eph_ind)## [1] "CODUSU_ind" "ANO4_ind" "TRIMESTRE_ind" "NRO_HOGAR_ind" ## [5] "COMPONENTE_ind" "H15_ind" "REGION_ind" "MAS_500_ind" ## [9] "AGLOMERADO_ind" "PONDERA_ind" "CH03_ind" "CH04_ind" ## [13] "CH05_ind" "CH06_ind" "CH07_ind" "CH08_ind" ## [17] "CH09_ind" "CH10_ind" "CH11_ind" "CH12_ind" ## [21] "CH13_ind" "CH14_ind" "CH15_ind" "CH15_COD_ind" ## [25] "CH16_ind" "CH16_COD_ind" "NIVEL_ED_ind" "ESTADO_ind" ## [29] "CAT_OCUP_ind" "CAT_INAC_ind" "IMPUTA_ind" "PP02C1_ind" ## [33] "PP02C2_ind" "PP02C3_ind" "PP02C4_ind" "PP02C5_ind" ## [37] "PP02C6_ind" "PP02C7_ind" "PP02C8_ind" "PP02E_ind" ## [41] "PP02H_ind" "PP02I_ind" "PP03C_ind" "PP03D_ind" ## [45] "PP3E_TOT_ind" "PP3F_TOT_ind" "PP03G_ind" "PP03H_ind" ## [49] "PP03I_ind" "PP03J_ind" "INTENSI_ind" "PP04A_ind" ## [53] "PP04B_COD_ind" "PP04B1_ind" "PP04B2_ind" "PP04B3_MES_ind"## [57] "PP04B3_ANO_ind" "PP04B3_DIA_ind" "PP04C_ind" "PP04C99_ind" ## [61] "PP04D_COD_ind" "PP04G_ind" "PP05B2_MES_ind" "PP05B2_ANO_ind"## [65] "PP05B2_DIA_ind" "PP05C_1_ind" "PP05C_2_ind" "PP05C_3_ind" ## [69] "PP05E_ind" "PP05F_ind" "PP05H_ind" "PP06A_ind" ## [73] "PP06C_ind" "PP06D_ind" "PP06E_ind" "PP06H_ind" ## [77] "PP07A_ind" "PP07C_ind" "PP07D_ind" "PP07E_ind" ## [81] "PP07F1_ind" "PP07F2_ind" "PP07F3_ind" "PP07F4_ind" ## [85] "PP07F5_ind" "PP07G1_ind" "PP07G2_ind" "PP07G3_ind" ## [89] "PP07G4_ind" "PP07G_59_ind" "PP07H_ind" "PP07I_ind" ## [93] "PP07J_ind" "PP07K_ind" "PP08D1_ind" "PP08D4_ind" ## [97] "PP08F1_ind" "PP08F2_ind" "PP08J1_ind" "PP08J2_ind" ## [101] "PP08J3_ind" "PP09A_ind" "PP09A_ESP_ind" "PP09B_ind" ## [105] "PP09C_ind" "PP09C_ESP_ind" "PP10A_ind" "PP10C_ind" ## [109] "PP10D_ind" "PP10E_ind" "PP11A_ind" "PP11B_COD_ind" ## [113] "PP11B1_ind" "PP11B2_MES_ind" "PP11B2_ANO_ind" "PP11B2_DIA_ind"## [117] "PP11C_ind" "PP11C99_ind" "PP11D_COD_ind" "PP11G_ANO_ind" ## [121] "PP11G_MES_ind" "PP11G_DIA_ind" "PP11L_ind" "PP11L1_ind" ## [125] "PP11M_ind" "PP11N_ind" "PP11O_ind" "PP11P_ind" ## [129] "PP11Q_ind" "PP11R_ind" "PP11S_ind" "PP11T_ind" ## [133] "P21_ind" "DECOCUR_ind" "IDECOCUR_ind" "RDECOCUR_ind" ## [137] "GDECOCUR_ind" "PDECOCUR_ind" "ADECOCUR_ind" "PONDIIO_ind" ## [141] "TOT_P12_ind" "P47T_ind" "DECINDR_ind" "IDECINDR_ind" ## [145] "RDECINDR_ind" "GDECINDR_ind" "PDECINDR_ind" "ADECINDR_ind" ## [149] "PONDII_ind" "V2_M_ind" "V3_M_ind" "V4_M_ind" ## [153] "V5_M_ind" "V8_M_ind" "V9_M_ind" "V10_M_ind" ## [157] "V11_M_ind" "V12_M_ind" "V18_M_ind" "V19_AM_ind" ## [161] "V21_M_ind" "T_VI_ind" "ITF_ind" "DECIFR_ind" ## [165] "IDECIFR_ind" "RDECIFR_ind" "GDECIFR_ind" "PDECIFR_ind" ## [169] "ADECIFR_ind" "IPCF_ind" "DECCFR_ind" "IDECCFR_ind" ## [173] "RDECCFR_ind" "GDECCFR_ind" "PDECCFR_ind" "ADECCFR_ind" ## [177] "PONDIH_ind"Por ahora, elijan creer
names(eph_ind)## [1] "CODUSU_ind" "ANO4_ind" "TRIMESTRE_ind" "NRO_HOGAR_ind" ## [5] "COMPONENTE_ind" "H15_ind" "REGION_ind" "MAS_500_ind" ## [9] "AGLOMERADO_ind" "PONDERA_ind" "CH03_ind" "CH04_ind" ## [13] "CH05_ind" "CH06_ind" "CH07_ind" "CH08_ind" ## [17] "CH09_ind" "CH10_ind" "CH11_ind" "CH12_ind" ## [21] "CH13_ind" "CH14_ind" "CH15_ind" "CH15_COD_ind" ## [25] "CH16_ind" "CH16_COD_ind" "NIVEL_ED_ind" "ESTADO_ind" ## [29] "CAT_OCUP_ind" "CAT_INAC_ind" "IMPUTA_ind" "PP02C1_ind" ## [33] "PP02C2_ind" "PP02C3_ind" "PP02C4_ind" "PP02C5_ind" ## [37] "PP02C6_ind" "PP02C7_ind" "PP02C8_ind" "PP02E_ind" ## [41] "PP02H_ind" "PP02I_ind" "PP03C_ind" "PP03D_ind" ## [45] "PP3E_TOT_ind" "PP3F_TOT_ind" "PP03G_ind" "PP03H_ind" ## [49] "PP03I_ind" "PP03J_ind" "INTENSI_ind" "PP04A_ind" ## [53] "PP04B_COD_ind" "PP04B1_ind" "PP04B2_ind" "PP04B3_MES_ind"## [57] "PP04B3_ANO_ind" "PP04B3_DIA_ind" "PP04C_ind" "PP04C99_ind" ## [61] "PP04D_COD_ind" "PP04G_ind" "PP05B2_MES_ind" "PP05B2_ANO_ind"## [65] "PP05B2_DIA_ind" "PP05C_1_ind" "PP05C_2_ind" "PP05C_3_ind" ## [69] "PP05E_ind" "PP05F_ind" "PP05H_ind" "PP06A_ind" ## [73] "PP06C_ind" "PP06D_ind" "PP06E_ind" "PP06H_ind" ## [77] "PP07A_ind" "PP07C_ind" "PP07D_ind" "PP07E_ind" ## [81] "PP07F1_ind" "PP07F2_ind" "PP07F3_ind" "PP07F4_ind" ## [85] "PP07F5_ind" "PP07G1_ind" "PP07G2_ind" "PP07G3_ind" ## [89] "PP07G4_ind" "PP07G_59_ind" "PP07H_ind" "PP07I_ind" ## [93] "PP07J_ind" "PP07K_ind" "PP08D1_ind" "PP08D4_ind" ## [97] "PP08F1_ind" "PP08F2_ind" "PP08J1_ind" "PP08J2_ind" ## [101] "PP08J3_ind" "PP09A_ind" "PP09A_ESP_ind" "PP09B_ind" ## [105] "PP09C_ind" "PP09C_ESP_ind" "PP10A_ind" "PP10C_ind" ## [109] "PP10D_ind" "PP10E_ind" "PP11A_ind" "PP11B_COD_ind" ## [113] "PP11B1_ind" "PP11B2_MES_ind" "PP11B2_ANO_ind" "PP11B2_DIA_ind"## [117] "PP11C_ind" "PP11C99_ind" "PP11D_COD_ind" "PP11G_ANO_ind" ## [121] "PP11G_MES_ind" "PP11G_DIA_ind" "PP11L_ind" "PP11L1_ind" ## [125] "PP11M_ind" "PP11N_ind" "PP11O_ind" "PP11P_ind" ## [129] "PP11Q_ind" "PP11R_ind" "PP11S_ind" "PP11T_ind" ## [133] "P21_ind" "DECOCUR_ind" "IDECOCUR_ind" "RDECOCUR_ind" ## [137] "GDECOCUR_ind" "PDECOCUR_ind" "ADECOCUR_ind" "PONDIIO_ind" ## [141] "TOT_P12_ind" "P47T_ind" "DECINDR_ind" "IDECINDR_ind" ## [145] "RDECINDR_ind" "GDECINDR_ind" "PDECINDR_ind" "ADECINDR_ind" ## [149] "PONDII_ind" "V2_M_ind" "V3_M_ind" "V4_M_ind" ## [153] "V5_M_ind" "V8_M_ind" "V9_M_ind" "V10_M_ind" ## [157] "V11_M_ind" "V12_M_ind" "V18_M_ind" "V19_AM_ind" ## [161] "V21_M_ind" "T_VI_ind" "ITF_ind" "DECIFR_ind" ## [165] "IDECIFR_ind" "RDECIFR_ind" "GDECIFR_ind" "PDECIFR_ind" ## [169] "ADECIFR_ind" "IPCF_ind" "DECCFR_ind" "IDECCFR_ind" ## [173] "RDECCFR_ind" "GDECCFR_ind" "PDECCFR_ind" "ADECCFR_ind" ## [177] "PONDIH_ind"
filter()select()rename()mutate()summarise()group_by()distinct()arrange()
Mutate ()
Mutate es la función que nos permite transformar las columnas, crear y modificarlas. En general, sin ser exhaustivos, podemos aplicar distintos tipos de transformaciones
Mutate ()
Mutate es la función que nos permite transformar las columnas, crear y modificarlas. En general, sin ser exhaustivos, podemos aplicar distintos tipos de transformaciones
- Operación aritmética sobre los valores numéricos
- Manipular caracteres en columnas de texto
- Una operación entre dos columnas
- Aplicarle cualquier función a la columna
Mutate ()
Mutate es la función que nos permite transformar las columnas, crear y modificarlas. En general, sin ser exhaustivos, podemos aplicar distintos tipos de transformaciones
- Operación aritmética sobre los valores numéricos
- Manipular caracteres en columnas de texto
- Una operación entre dos columnas
- Aplicarle cualquier función a la columna
En todos los casos podemos crear una columna nueva o modificar la que ya existe
Mutate ()
Mutate es la función que nos permite transformar las columnas, crear y modificarlas. En general, sin ser exhaustivos, podemos aplicar distintos tipos de transformaciones
- Operación aritmética sobre los valores numéricos
- Manipular caracteres en columnas de texto
- Una operación entre dos columnas
- Aplicarle cualquier función a la columna
En todos los casos podemos crear una columna nueva o modificar la que ya existe
Por ejemplo, si quisiéramos crear una columna con el IPCF en dolares:
eph_ind <- eph_ind %>% mutate(IPCF_USD = as.numeric(IPCF)/390)Mutate ()
Mutate es la función que nos permite transformar las columnas, crear y modificarlas. En general, sin ser exhaustivos, podemos aplicar distintos tipos de transformaciones
- Operación aritmética sobre los valores numéricos
- Manipular caracteres en columnas de texto
- Una operación entre dos columnas
- Aplicarle cualquier función a la columna
En todos los casos podemos crear una columna nueva o modificar la que ya existe
Por ejemplo, si quisiéramos crear una columna con el IPCF en dolares:
eph_ind <- eph_ind %>% mutate(IPCF_USD = as.numeric(IPCF)/390)Notar que aquí aplicamos dos transformaciones, primero as.numeric() y luego dividimos por el tipo de cambio
Mutate ()
Si queremos sumar los distintos ingresos laborales que puede tener una persona:
- Monto de ingreso de la ocupación principal (P21)
- Monto de ingreso de otras ocupaciones (TOT_P12)
Mutate ()
Si queremos sumar los distintos ingresos laborales que puede tener una persona:
- Monto de ingreso de la ocupación principal (P21)
- Monto de ingreso de otras ocupaciones (TOT_P12)
eph_ind <- eph_ind %>% mutate(ILA_TOT = P21 + TOT_P12)Mutate ()
Si queremos sumar los distintos ingresos laborales que puede tener una persona:
- Monto de ingreso de la ocupación principal (P21)
- Monto de ingreso de otras ocupaciones (TOT_P12)
eph_ind <- eph_ind %>% mutate(ILA_TOT = P21 + TOT_P12)Si queremos trabajar con el logaritmo del ingreso de la ocupación principal, debemos crear la columna mediante la función log()
eph_ind <- eph_ind %>% mutate(log_w = log(P21))Mutate ()
Para el análisis de datos, muchas veces necesitamos crear variables binarias a partir de otras variables en la base
Mutate ()
Para el análisis de datos, muchas veces necesitamos crear variables binarias a partir de otras variables en la base
Por ejemplo, en la EPH tenemos la variable MAS_500 que indica si el aglomerada tiene mas de 500.000 habitantes
table(eph_ind$MAS_500)## ## N S ## 26120 23112Mutate ()
Para el análisis de datos, muchas veces necesitamos crear variables binarias a partir de otras variables en la base
Por ejemplo, en la EPH tenemos la variable MAS_500 que indica si el aglomerada tiene mas de 500.000 habitantes
table(eph_ind$MAS_500)## ## N S ## 26120 23112Parece ser que es del tipo character
is.character(eph_ind$MAS_500)## [1] TRUEMutate ()
Supongamos que con ese umbral de 500 mil, definiremos un aglomerado como urbano o no, y para eso debemos crear una variable nueva, llamada URBANO
Mutate ()
Supongamos que con ese umbral de 500 mil, definiremos un aglomerado como urbano o no, y para eso debemos crear una variable nueva, llamada URBANO
Necesitamos un función que contemple condiciones:
- si MAS_500=="N" ; URBANO=0
- si MAS_500=="S" ; URBANO=1
Mutate ()
Supongamos que con ese umbral de 500 mil, definiremos un aglomerado como urbano o no, y para eso debemos crear una variable nueva, llamada URBANO
Necesitamos un función que contemple condiciones:
- si MAS_500=="N" ; URBANO=0
- si MAS_500=="S" ; URBANO=1
ifelse() hace perfectamente este trabajo
eph_ind <- eph_ind %>% mutate(URBANO = ifelse(MAS_500=="S", 1, 0))Entonces ahora puedo saber qué proporción de los encuestados vive en lo que definimos como zona urbana
mean(eph_ind$URBANO)## [1] 0.4694508Entonces ahora puedo saber qué proporción de los encuestados vive en lo que definimos como zona urbana
mean(eph_ind$URBANO)## [1] 0.4694508x <- round( mean(eph_ind$URBANO), digits=2)*100print(paste("el ", x, "% de las personas viven en zona urbana", sep=""))## [1] "el 47% de las personas viven en zona urbana"Mutate ()
¿Qué ocurre en casos donde la cantidad de condiciones posibles son mas de 2?
Por ejemplo, en el caso de la variable región
table(eph_ind$REGION)## ## 1 40 41 42 43 44 ## 7344 10842 5153 5183 14732 5978Aquí necesitamos de la función case_when(). Como su nombre lo indica, lo lógica es: si pasa esto, tal cosa, si pasa esto otro tal otra y así las veces que necesitemos
Creemos una nueva variable indicativa de la región pero que ahora sea character
eph_ind <- eph_ind %>% mutate(REGION_CH = case_when(REGION==1 ~ "Gran Buenos Aires", REGION==40 ~ "Noroeste", REGION==41 ~ "Nordeste", REGION==42 ~ "Cuyo", REGION==43 ~ "Pampeana", REGION==44 ~ "Patagónica")) #DOBLE()Creemos una nueva variable indicativa de la región pero que ahora sea character
eph_ind <- eph_ind %>% mutate(REGION_CH = case_when(REGION==1 ~ "Gran Buenos Aires", REGION==40 ~ "Noroeste", REGION==41 ~ "Nordeste", REGION==42 ~ "Cuyo", REGION==43 ~ "Pampeana", REGION==44 ~ "Patagónica")) #DOBLE()table(eph_ind$REGION_CH)## ## Cuyo Gran Buenos Aires Nordeste Noroeste ## 5183 7344 5153 10842 ## Pampeana Patagónica ## 14732 5978Creemos una nueva variable indicativa de la región pero que ahora sea character
eph_ind <- eph_ind %>% mutate(REGION_CH = case_when(REGION==1 ~ "Gran Buenos Aires", REGION==40 ~ "Noroeste", REGION==41 ~ "Nordeste", REGION==42 ~ "Cuyo", REGION==43 ~ "Pampeana", REGION==44 ~ "Patagónica")) #DOBLE()table(eph_ind$REGION_CH)## ## Cuyo Gran Buenos Aires Nordeste Noroeste ## 5183 7344 5153 10842 ## Pampeana Patagónica ## 14732 5978Nuevamente, es bueno visualizar lo que ocurre en la base de datos para entender mejor las transformaciones que realizamos
Empezamos llamando al data frame eph_ind, seleccionamos columnas de interés y filtramos a los ocupados
ejemplo <- eph_ind %>% select(CODUSU, AGLOMERADO, ESTADO, P21, REGION) %>% filter(ESTADO==1)| CODUSU | AGLOMERADO | ESTADO | P21 | REGION |
|---|---|---|---|---|
| TQRMNORVQHJNLTCDEGGFB00788741 | 10 | 1 | 20000 | 42 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 29 | 1 | 0 | 40 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 29 | 1 | 0 | 40 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 91 | 1 | 168000 | 44 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 7 | 1 | 70000 | 41 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 7 | 1 | 30000 | 41 |
| TQRMNOPXUHLMKUCDEGOIH00706727 | 18 | 1 | 80000 | 40 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 12 | 1 | 99000 | 41 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 12 | 1 | 12000 | 41 |
| TQRMNOQSUHMLKTCDEHIBB00717071 | 22 | 1 | 80000 | 40 |
| TQRMNOQSUHMLKTCDEHIBB00717071 | 22 | 1 | 115000 | 40 |
| TQRMNOQSQHMLKTCDEHIBB00709425 | 22 | 1 | 96000 | 40 |
| TQRMNOQTVHJMKTCDEHIBB00784254 | 22 | 1 | 100000 | 40 |
| TQRMNOQTVHJMKTCDEHIBB00784254 | 22 | 1 | 25000 | 40 |
| TQRMNOQXUHLMRQCDEIJAH00706181 | 33 | 1 | 44000 | 1 |
| TQRMNORPQHLMKSCDEHJGH00790306 | 23 | 1 | -9 | 40 |
| TQRMNOQXUHMLSNCDEIJAH00719506 | 33 | 1 | 73000 | 1 |
| TQRMNORPUHJNOLCDEIIAD00785503 | 32 | 1 | 110000 | 1 |
| TQRMNORQRHJLKNCDEFMDB00782419 | 6 | 1 | 100000 | 43 |
| TQRMNOQWUHJMKUCDEHGHJ00784122 | 20 | 1 | 90000 | 44 |
| TQRMNOPVSHMNQMCDEIJAH00792376 | 33 | 1 | -9 | 1 |
| TQRMNOQUYHMNKPCDEIOAH00710452 | 38 | 1 | 100000 | 43 |
| TQRMNOQUYHMNKPCDEIOAH00710452 | 38 | 1 | 10000 | 43 |
| TQRMNOSUTHJMKTCDEHIBB00784269 | 22 | 1 | 65000 | 40 |
| TQRMNOSUTHJMKTCDEHIBB00784269 | 22 | 1 | 25000 | 40 |
| TQRMNORWQHLOLQCDEIIAD00791536 | 32 | 1 | 70000 | 1 |
| TQRMNOPPQHJLONCDEIIAD00785511 | 32 | 1 | -9 | 1 |
| TQRMNOPPQHJLONCDEIIAD00785511 | 32 | 1 | -9 | 1 |
| TQRMNORQTHLOSNCDEIJAH00711164 | 33 | 1 | 40000 | 1 |
| TQSMNOQUWHMLMNCDEGJBF00789124 | 13 | 1 | 36000 | 43 |
| TQRMNOPTTHJMLNCDEGGFB00783107 | 10 | 1 | -9 | 42 |
| TQRMNOPTQHKMKQCDEGPDJ00789889 | 19 | 1 | 18000 | 40 |
| TQRMNOSPXHKOLQCDEGNFJ00789612 | 17 | 1 | 12000 | 44 |
| TQRMNOUPYHKOLQCDEGNFJ00789613 | 17 | 1 | 0 | 44 |
| TQRMNOUPYHKOLQCDEGNFJ00789613 | 17 | 1 | 90000 | 44 |
| TQRMNOPQVHMOLPCDEFLID00715014 | 5 | 1 | -9 | 43 |
| TQRMNOVQTHJOPSCDEIJAH00792281 | 33 | 1 | 60000 | 1 |
| TQRMNOPQXHMOLPCDEFLID00710841 | 5 | 1 | 60000 | 43 |
| TQRMNOPQXHMOLPCDEFLID00710841 | 5 | 1 | 85000 | 43 |
| TQRMNOSTPHLMLLCDEGKDB00705046 | 14 | 1 | 60000 | 43 |
| TQRMNOSTPHLMLLCDEGKDB00705046 | 14 | 1 | -9 | 43 |
| TQRMNOSTPHLMLLCDEGKDB00705046 | 14 | 1 | -9 | 43 |
| TQRMNOPYSHKOKQCDEFLID00787875 | 5 | 1 | 30000 | 43 |
| TQRMNORTPHKOKQCDEFLID00787876 | 5 | 1 | 40000 | 43 |
| TQRMNORSPHMMPMCDEIJAH00709078 | 33 | 1 | 70000 | 1 |
| TQRMNOQQWHKOKRCDEGOIH00789766 | 18 | 1 | 35000 | 40 |
| TQRMNOQQWHKOKRCDEGOIH00789766 | 18 | 1 | 12000 | 40 |
| TQRMNORRVHMOKRCDEOJAH00713918 | 93 | 1 | 110000 | 44 |
| TQRMNORRVHMOKRCDEOJAH00713918 | 93 | 1 | 85000 | 44 |
| TQRMNOQUQHLOKSCDEGOIH00706615 | 18 | 1 | 70000 | 40 |
(aquí imprimiremos a modo ilustrativo solo las primeras 50 filas)
Convertimos todo a minúscula y renombramos la variable P21
ejemplo <- eph_ind %>% select(CODUSU, AGLOMERADO, ESTADO, P21, REGION) %>% filter(ESTADO==1) %>% rename_with(tolower) %>% rename(inglab = p21)| codusu | aglomerado | estado | inglab | region |
|---|---|---|---|---|
| TQRMNORVQHJNLTCDEGGFB00788741 | 10 | 1 | 20000 | 42 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 29 | 1 | 0 | 40 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 29 | 1 | 0 | 40 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 91 | 1 | 168000 | 44 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 7 | 1 | 70000 | 41 |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 7 | 1 | 30000 | 41 |
| TQRMNOPXUHLMKUCDEGOIH00706727 | 18 | 1 | 80000 | 40 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 12 | 1 | 99000 | 41 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 12 | 1 | 12000 | 41 |
| TQRMNOQSUHMLKTCDEHIBB00717071 | 22 | 1 | 80000 | 40 |
| TQRMNOQSUHMLKTCDEHIBB00717071 | 22 | 1 | 115000 | 40 |
| TQRMNOQSQHMLKTCDEHIBB00709425 | 22 | 1 | 96000 | 40 |
| TQRMNOQTVHJMKTCDEHIBB00784254 | 22 | 1 | 100000 | 40 |
| TQRMNOQTVHJMKTCDEHIBB00784254 | 22 | 1 | 25000 | 40 |
| TQRMNOQXUHLMRQCDEIJAH00706181 | 33 | 1 | 44000 | 1 |
| TQRMNORPQHLMKSCDEHJGH00790306 | 23 | 1 | -9 | 40 |
| TQRMNOQXUHMLSNCDEIJAH00719506 | 33 | 1 | 73000 | 1 |
| TQRMNORPUHJNOLCDEIIAD00785503 | 32 | 1 | 110000 | 1 |
| TQRMNORQRHJLKNCDEFMDB00782419 | 6 | 1 | 100000 | 43 |
| TQRMNOQWUHJMKUCDEHGHJ00784122 | 20 | 1 | 90000 | 44 |
| TQRMNOPVSHMNQMCDEIJAH00792376 | 33 | 1 | -9 | 1 |
| TQRMNOQUYHMNKPCDEIOAH00710452 | 38 | 1 | 100000 | 43 |
| TQRMNOQUYHMNKPCDEIOAH00710452 | 38 | 1 | 10000 | 43 |
| TQRMNOSUTHJMKTCDEHIBB00784269 | 22 | 1 | 65000 | 40 |
| TQRMNOSUTHJMKTCDEHIBB00784269 | 22 | 1 | 25000 | 40 |
| TQRMNORWQHLOLQCDEIIAD00791536 | 32 | 1 | 70000 | 1 |
| TQRMNOPPQHJLONCDEIIAD00785511 | 32 | 1 | -9 | 1 |
| TQRMNOPPQHJLONCDEIIAD00785511 | 32 | 1 | -9 | 1 |
| TQRMNORQTHLOSNCDEIJAH00711164 | 33 | 1 | 40000 | 1 |
| TQSMNOQUWHMLMNCDEGJBF00789124 | 13 | 1 | 36000 | 43 |
| TQRMNOPTTHJMLNCDEGGFB00783107 | 10 | 1 | -9 | 42 |
| TQRMNOPTQHKMKQCDEGPDJ00789889 | 19 | 1 | 18000 | 40 |
| TQRMNOSPXHKOLQCDEGNFJ00789612 | 17 | 1 | 12000 | 44 |
| TQRMNOUPYHKOLQCDEGNFJ00789613 | 17 | 1 | 0 | 44 |
| TQRMNOUPYHKOLQCDEGNFJ00789613 | 17 | 1 | 90000 | 44 |
| TQRMNOPQVHMOLPCDEFLID00715014 | 5 | 1 | -9 | 43 |
| TQRMNOVQTHJOPSCDEIJAH00792281 | 33 | 1 | 60000 | 1 |
| TQRMNOPQXHMOLPCDEFLID00710841 | 5 | 1 | 60000 | 43 |
| TQRMNOPQXHMOLPCDEFLID00710841 | 5 | 1 | 85000 | 43 |
| TQRMNOSTPHLMLLCDEGKDB00705046 | 14 | 1 | 60000 | 43 |
| TQRMNOSTPHLMLLCDEGKDB00705046 | 14 | 1 | -9 | 43 |
| TQRMNOSTPHLMLLCDEGKDB00705046 | 14 | 1 | -9 | 43 |
| TQRMNOPYSHKOKQCDEFLID00787875 | 5 | 1 | 30000 | 43 |
| TQRMNORTPHKOKQCDEFLID00787876 | 5 | 1 | 40000 | 43 |
| TQRMNORSPHMMPMCDEIJAH00709078 | 33 | 1 | 70000 | 1 |
| TQRMNOQQWHKOKRCDEGOIH00789766 | 18 | 1 | 35000 | 40 |
| TQRMNOQQWHKOKRCDEGOIH00789766 | 18 | 1 | 12000 | 40 |
| TQRMNORRVHMOKRCDEOJAH00713918 | 93 | 1 | 110000 | 44 |
| TQRMNORRVHMOKRCDEOJAH00713918 | 93 | 1 | 85000 | 44 |
| TQRMNOQUQHLOKSCDEGOIH00706615 | 18 | 1 | 70000 | 40 |
Generamos una columna para el log del ingreso y un para región como character
ejemplo <- eph_ind %>% select(CODUSU, AGLOMERADO, ESTADO, P21, REGION)%>% filter(ESTADO==1) %>% rename_with(tolower) %>% rename(inglab = p21) %>% mutate(log_inglab = log(inglab), region_ch = case_when(region==1 ~ "Gran Buenos Aires", region==40 ~ "Noroeste", region==41 ~ "Nordeste", region==42 ~ "Cuyo", region==43 ~ "Pampeana", region==44 ~ "Patagónica")) #DOBLE()| codusu | aglomerado | estado | inglab | region | log_inglab | region_ch |
|---|---|---|---|---|---|---|
| TQRMNORVQHJNLTCDEGGFB00788741 | 10 | 1 | 20000 | 42 | 9.903488 | Cuyo |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 29 | 1 | 0 | 40 | -Inf | Noroeste |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 29 | 1 | 0 | 40 | -Inf | Noroeste |
| TQRMNORRTHMMLLCDEOHCH00710640 | 91 | 1 | 168000 | 44 | 12.031719 | Patagónica |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 7 | 1 | 70000 | 41 | 11.156251 | Nordeste |
| TQRMNOQVXHMOKUCDEFNFF00710948 | 7 | 1 | 30000 | 41 | 10.308953 | Nordeste |
| TQRMNOPXUHLMKUCDEGOIH00706727 | 18 | 1 | 80000 | 40 | 11.289782 | Noroeste |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 12 | 1 | 99000 | 41 | 11.502875 | Nordeste |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 12 | 1 | 12000 | 41 | 9.392662 | Nordeste |
| TQRMNOQSUHMLKTCDEHIBB00717071 | 22 | 1 | 80000 | 40 | 11.289782 | Noroeste |
| TQRMNOQSUHMLKTCDEHIBB00717071 | 22 | 1 | 115000 | 40 | 11.652687 | Noroeste |
| TQRMNOQSQHMLKTCDEHIBB00709425 | 22 | 1 | 96000 | 40 | 11.472103 | Noroeste |
| TQRMNOQTVHJMKTCDEHIBB00784254 | 22 | 1 | 100000 | 40 | 11.512925 | Noroeste |
| TQRMNOQTVHJMKTCDEHIBB00784254 | 22 | 1 | 25000 | 40 | 10.126631 | Noroeste |
| TQRMNOQXUHLMRQCDEIJAH00706181 | 33 | 1 | 44000 | 1 | 10.691945 | Gran Buenos Aires |
| TQRMNORPQHLMKSCDEHJGH00790306 | 23 | 1 | -9 | 40 | NaN | Noroeste |
| TQRMNOQXUHMLSNCDEIJAH00719506 | 33 | 1 | 73000 | 1 | 11.198215 | Gran Buenos Aires |
| TQRMNORPUHJNOLCDEIIAD00785503 | 32 | 1 | 110000 | 1 | 11.608236 | Gran Buenos Aires |
| TQRMNORQRHJLKNCDEFMDB00782419 | 6 | 1 | 100000 | 43 | 11.512925 | Pampeana |
| TQRMNOQWUHJMKUCDEHGHJ00784122 | 20 | 1 | 90000 | 44 | 11.407565 | Patagónica |
| TQRMNOPVSHMNQMCDEIJAH00792376 | 33 | 1 | -9 | 1 | NaN | Gran Buenos Aires |
| TQRMNOQUYHMNKPCDEIOAH00710452 | 38 | 1 | 100000 | 43 | 11.512925 | Pampeana |
| TQRMNOQUYHMNKPCDEIOAH00710452 | 38 | 1 | 10000 | 43 | 9.210340 | Pampeana |
| TQRMNOSUTHJMKTCDEHIBB00784269 | 22 | 1 | 65000 | 40 | 11.082142 | Noroeste |
| TQRMNOSUTHJMKTCDEHIBB00784269 | 22 | 1 | 25000 | 40 | 10.126631 | Noroeste |
| TQRMNORWQHLOLQCDEIIAD00791536 | 32 | 1 | 70000 | 1 | 11.156251 | Gran Buenos Aires |
| TQRMNOPPQHJLONCDEIIAD00785511 | 32 | 1 | -9 | 1 | NaN | Gran Buenos Aires |
| TQRMNOPPQHJLONCDEIIAD00785511 | 32 | 1 | -9 | 1 | NaN | Gran Buenos Aires |
| TQRMNORQTHLOSNCDEIJAH00711164 | 33 | 1 | 40000 | 1 | 10.596635 | Gran Buenos Aires |
| TQSMNOQUWHMLMNCDEGJBF00789124 | 13 | 1 | 36000 | 43 | 10.491274 | Pampeana |
| TQRMNOPTTHJMLNCDEGGFB00783107 | 10 | 1 | -9 | 42 | NaN | Cuyo |
| TQRMNOPTQHKMKQCDEGPDJ00789889 | 19 | 1 | 18000 | 40 | 9.798127 | Noroeste |
| TQRMNOSPXHKOLQCDEGNFJ00789612 | 17 | 1 | 12000 | 44 | 9.392662 | Patagónica |
| TQRMNOUPYHKOLQCDEGNFJ00789613 | 17 | 1 | 0 | 44 | -Inf | Patagónica |
| TQRMNOUPYHKOLQCDEGNFJ00789613 | 17 | 1 | 90000 | 44 | 11.407565 | Patagónica |
| TQRMNOPQVHMOLPCDEFLID00715014 | 5 | 1 | -9 | 43 | NaN | Pampeana |
| TQRMNOVQTHJOPSCDEIJAH00792281 | 33 | 1 | 60000 | 1 | 11.002100 | Gran Buenos Aires |
| TQRMNOPQXHMOLPCDEFLID00710841 | 5 | 1 | 60000 | 43 | 11.002100 | Pampeana |
| TQRMNOPQXHMOLPCDEFLID00710841 | 5 | 1 | 85000 | 43 | 11.350407 | Pampeana |
| TQRMNOSTPHLMLLCDEGKDB00705046 | 14 | 1 | 60000 | 43 | 11.002100 | Pampeana |
| TQRMNOSTPHLMLLCDEGKDB00705046 | 14 | 1 | -9 | 43 | NaN | Pampeana |
| TQRMNOSTPHLMLLCDEGKDB00705046 | 14 | 1 | -9 | 43 | NaN | Pampeana |
| TQRMNOPYSHKOKQCDEFLID00787875 | 5 | 1 | 30000 | 43 | 10.308953 | Pampeana |
| TQRMNORTPHKOKQCDEFLID00787876 | 5 | 1 | 40000 | 43 | 10.596635 | Pampeana |
| TQRMNORSPHMMPMCDEIJAH00709078 | 33 | 1 | 70000 | 1 | 11.156251 | Gran Buenos Aires |
| TQRMNOQQWHKOKRCDEGOIH00789766 | 18 | 1 | 35000 | 40 | 10.463103 | Noroeste |
| TQRMNOQQWHKOKRCDEGOIH00789766 | 18 | 1 | 12000 | 40 | 9.392662 | Noroeste |
| TQRMNORRVHMOKRCDEOJAH00713918 | 93 | 1 | 110000 | 44 | 11.608236 | Patagónica |
| TQRMNORRVHMOKRCDEOJAH00713918 | 93 | 1 | 85000 | 44 | 11.350407 | Patagónica |
| TQRMNOQUQHLOKSCDEGOIH00706615 | 18 | 1 | 70000 | 40 | 11.156251 | Noroeste |
filter()select()rename()mutate()summarise()group_by()distinct()arrange()
Summarise()
Función por excelencia para generar medidas resúmenes de las observaciones de datos.
Summarise()
Función por excelencia para generar medidas resúmenes de las observaciones de datos.
Colapsa la base a un valor o valores según la operación aplicada.
Summarise()
Función por excelencia para generar medidas resúmenes de las observaciones de datos.
Colapsa la base a un valor o valores según la operación aplicada.
Retomando nuestro ejemplo inicial, supongamos que quisiéramos obtener la media de edad y la mediana del ingreso a nivel país
resumen <- eph_ind %>% filter(!is.na(IPCF)) %>% rename(sexo = CH04, edad = CH06) %>% rename_with(tolower) %>% mutate(ipcf = as.numeric(ipcf)) %>% summarise(edad_mean = mean(edad, na.rm=TRUE), ipcf_median = median(ipcf, na.rm = TRUE))Summarise()
Notar que antes, como resultados obteníamos un dataframe, es decir filtramos las filas, nos quedábamos con menos columnas y las modificábamos, pero el resultado seguía siendo una base tabular.
Summarise()
Notar que antes, como resultados obteníamos un dataframe, es decir filtramos las filas, nos quedábamos con menos columnas y las modificábamos, pero el resultado seguía siendo una base tabular.
Ahora summarise() colapsa todos las observaciones a un único valor o valores
Summarise()
Notar que antes, como resultados obteníamos un dataframe, es decir filtramos las filas, nos quedábamos con menos columnas y las modificábamos, pero el resultado seguía siendo una base tabular.
Ahora summarise() colapsa todos las observaciones a un único valor o valores
resumen## edad_mean ipcf_median## 1 36.00443 27500Summarise()
Notar que antes, como resultados obteníamos un dataframe, es decir filtramos las filas, nos quedábamos con menos columnas y las modificábamos, pero el resultado seguía siendo una base tabular.
Ahora summarise() colapsa todos las observaciones a un único valor o valores
resumen## edad_mean ipcf_median## 1 36.00443 27500Otro ejemplo, para los ocupados calculemos el promedio y el máximo de horas trabajadas en la ocupación principal (PP3E_TOT)
eph_ind %>% filter(ESTADO==1) %>% summarise(mean_hs = mean(PP3E_TOT), max_hs = max(PP3E_TOT))## mean_hs max_hs## 1 36.85635 999Summarise() vs Mutate()
Cuál es la diferencia entre estas dos funciones? Al parecer ambas sirven para generar nuevas columnas o reemplazarlas por nuevos valores
Summarise() vs Mutate()
Cuál es la diferencia entre estas dos funciones? Al parecer ambas sirven para generar nuevas columnas o reemplazarlas por nuevos valores
mutate()modifica una o varias columnas pero no altera la estructura de la base
Summarise() vs Mutate()
Cuál es la diferencia entre estas dos funciones? Al parecer ambas sirven para generar nuevas columnas o reemplazarlas por nuevos valores
mutate()modifica una o varias columnas pero no altera la estructura de la base

Es del tipo Window Function que toma un vector de valores y devuelve otro vector de valores

Summarise() vs Mutate()
summarise()genera una columna o reemplaza una existente colapsando las base (cambia las dimensiones)
Summarise() vs Mutate()
summarise()genera una columna o reemplaza una existente colapsando las base (cambia las dimensiones)

Es del tipo Summary Function que toma un vector de valores y devuelve un valor único

Summarise() vs Mutate()
El ejemplo anterior nos puede servir. Con el max de horas vimos que existen valores raros o imposibles, estos nos afectan el calculo del promedio, de la media, varianza, etc.
Summarise() vs Mutate()
El ejemplo anterior nos puede servir. Con el max de horas vimos que existen valores raros o imposibles, estos nos afectan el calculo del promedio, de la media, varianza, etc.
Tenemos que corregir ese valor antes de generar medidas resúmenes.
Summarise() vs Mutate()
El ejemplo anterior nos puede servir. Con el max de horas vimos que existen valores raros o imposibles, estos nos afectan el calculo del promedio, de la media, varianza, etc.
Tenemos que corregir ese valor antes de generar medidas resúmenes.
¿Se les ocurre cómo?
Summarise() vs Mutate()
resumen <- eph_ind %>% filter(ESTADO==1) %>% mutate(PP3E_TOT = ifelse(PP3E_TOT==999, NA, PP3E_TOT))mutate() genera un cambio en la columna sin alterar la estructura de la base:
Summarise() vs Mutate()
resumen <- eph_ind %>% filter(ESTADO==1) %>% mutate(PP3E_TOT = ifelse(PP3E_TOT==999, NA, PP3E_TOT))mutate() genera un cambio en la columna sin alterar la estructura de la base:
dim(resumen)## [1] 21364 182Summarise() vs Mutate()
resumen <- eph_ind %>% filter(ESTADO==1) %>% mutate(PP3E_TOT = ifelse(PP3E_TOT==999, NA, PP3E_TOT))mutate() genera un cambio en la columna sin alterar la estructura de la base:
dim(resumen)## [1] 21364 182Ahora genero medida resumen
resumen <- resumen %>% summarise(mean_hs = mean(PP3E_TOT), max_hs = max(PP3E_TOT))Summarise() vs Mutate()
resumen <- eph_ind %>% filter(ESTADO==1) %>% mutate(PP3E_TOT = ifelse(PP3E_TOT==999, NA, PP3E_TOT))mutate() genera un cambio en la columna sin alterar la estructura de la base:
dim(resumen)## [1] 21364 182Ahora genero medida resumen
resumen <- resumen %>% summarise(mean_hs = mean(PP3E_TOT), max_hs = max(PP3E_TOT))dim(resumen)## [1] 1 2summarise() colapsó la base original
¿Cuál fue el resultado de esa operación?
resumen## mean_hs max_hs## 1 NA NAQue pasó?
¿Cuál fue el resultado de esa operación?
resumen## mean_hs max_hs## 1 NA NAQue pasó?
Exacto! Ahora introdujimos NA en la columna y nos olvidamos de pedirle a la función que los ignore
¿Cuál fue el resultado de esa operación?
resumen## mean_hs max_hs## 1 NA NAQue pasó?
Exacto! Ahora introdujimos NA en la columna y nos olvidamos de pedirle a la función que los ignore
resumen <- eph_ind %>% filter(ESTADO==1) %>% mutate(PP3E_TOT = ifelse(PP3E_TOT==999, NA, PP3E_TOT)) %>% summarise(mean_hs = mean(PP3E_TOT, na.rm=TRUE), max_hs = max(PP3E_TOT, na.rm=TRUE))¿Cuál fue el resultado de esa operación?
resumen## mean_hs max_hs## 1 NA NAQue pasó?
Exacto! Ahora introdujimos NA en la columna y nos olvidamos de pedirle a la función que los ignore
resumen <- eph_ind %>% filter(ESTADO==1) %>% mutate(PP3E_TOT = ifelse(PP3E_TOT==999, NA, PP3E_TOT)) %>% summarise(mean_hs = mean(PP3E_TOT, na.rm=TRUE), max_hs = max(PP3E_TOT, na.rm=TRUE))No solo cambió el calculo del máximo, al quitar los valores raros también cambia la estimación de la media
Summarise()
En nuestro ejemplo, solo empleamos la media, mediana y el máximo, pero la lista de funciones útiles para aplicar con summarise() es mucho mas extensa
Veamos las más comunes
Center:
mean(),median()Spread:
sd(),IQR(),mad()Range:
min(),max()Position:
first(),last(),nth()Count:
n(),n_distinct()Logical:
any(),all()
filter()select()rename()mutate()summarise()group_by()distinct()arrange()
Group by()
Aún nos queda por ver el uso más común que se le suele dar al summarise()
Group by()
Aún nos queda por ver el uso más común que se le suele dar al summarise()Para ello tenemos que introducir antes a group_by()
Group by()
Aún nos queda por ver el uso más común que se le suele dar al summarise()Para ello tenemos que introducir antes a group_by()
Rara vez reducimos todas las observaciones en una sola dimensión. En general en el análisis económico nos interesan las diferencias entre distintos grupos, buscamos agrupar las observaciones para compararlas, encontrar patrones que nos permitan hablar de heterogeneidades, etc.
Group by()
Aún nos queda por ver el uso más común que se le suele dar al summarise()Para ello tenemos que introducir antes a group_by()
Rara vez reducimos todas las observaciones en una sola dimensión. En general en el análisis económico nos interesan las diferencias entre distintos grupos, buscamos agrupar las observaciones para compararlas, encontrar patrones que nos permitan hablar de heterogeneidades, etc.
Cómo pueden sospechar, group_by() nos habilita este tipo de análisis. Como función nos permite realizar una misma operación para un conjunto o grupos de observaciones.
Group by() + Summarise()

Group by() + Summarise()

Al agrupar las observaciones por alguna o varias dimensiones en común, estaremos tratando a cada conjunto como una unidad aparte.
Group by() + Summarise()
Por ejemplo, si en nuestro ejemplo ahora quisiéramos ver cuál es la diferencia en la mediana del salario y en el promedio de horas trabajadas por género
Group by() + Summarise()
Por ejemplo, si en nuestro ejemplo ahora quisiéramos ver cuál es la diferencia en la mediana del salario y en el promedio de horas trabajadas por género
subset <- eph_ind %>% filter(!is.na(IPCF)) %>% rename(sexo = CH04, edad = CH06) %>% rename_with(tolower) %>% mutate(ipcf = as.numeric(ipcf), pp3e_tot = ifelse(pp3e_tot==999, NA, pp3e_tot))gender_diff <- subset %>% group_by(sexo) %>% summarise(mean_hs = mean(pp3e_tot, na.rm=TRUE), ipcf_median = median(ipcf, na.rm = TRUE))View(gender_diff)View(gender_diff)| sexo | mean_hs | ipcf_median |
|---|---|---|
| 1 | 39.63996 | 27672.73 |
| 2 | 30.73663 | 27200.00 |
View(gender_diff)| sexo | mean_hs | ipcf_median |
|---|---|---|
| 1 | 39.63996 | 27672.73 |
| 2 | 30.73663 | 27200.00 |
Ahora la cantidad de filas resultantes son igual a la cantidad de grupos distintos que surgen del group_by()
Group by() + Summarise()
El agrupamiento puede ser por mas de una variable
Por ejemplo, si queremos saber si la diferencia en medias salariales y en horas trabajadas es distintas en zonas urbanos y no urbanas, deberíamos agrupar por ambas variables
Group by() + Summarise()
El agrupamiento puede ser por mas de una variable
Por ejemplo, si queremos saber si la diferencia en medias salariales y en horas trabajadas es distintas en zonas urbanos y no urbanas, deberíamos agrupar por ambas variables
gender_diff_ur <- subset %>% mutate(URBANO = ifelse(mas_500=="S", 1, 0)) %>% group_by(sexo, urbano) %>% summarise(mean_hs = mean(pp3e_tot, na.rm=TRUE), ipcf_median = median(ipcf, na.rm = TRUE))Group by() + Summarise()
El agrupamiento puede ser por mas de una variable
Por ejemplo, si queremos saber si la diferencia en medias salariales y en horas trabajadas es distintas en zonas urbanos y no urbanas, deberíamos agrupar por ambas variables
gender_diff_ur <- subset %>% mutate(URBANO = ifelse(mas_500=="S", 1, 0)) %>% group_by(sexo, urbano) %>% summarise(mean_hs = mean(pp3e_tot, na.rm=TRUE), ipcf_median = median(ipcf, na.rm = TRUE))| sexo | urbano | mean_hs | ipcf_median |
|---|---|---|---|
| 1 | 0 | 40.09855 | 29000.00 |
| 1 | 1 | 39.14594 | 26500.00 |
| 2 | 0 | 31.14342 | 28333.33 |
| 2 | 1 | 30.31133 | 25600.00 |
Group by() + Mutate()
Además de calcular estimaciones por grupos, el agrupamiento es útil cuando lo combinamos con la transformación de variables, antes de colapsar la base
Group by() + Mutate()
Por ejemplo si quisiéramos saber la distribución de hogares según la edad del hijo menor, porque nos interesa conocer cuantos hogares serían alcanzados por una política focalizada en la nutrición de niños hasta 5 años
Group by() + Mutate()
Por ejemplo si quisiéramos saber la distribución de hogares según la edad del hijo menor, porque nos interesa conocer cuantos hogares serían alcanzados por una política focalizada en la nutrición de niños hasta 5 años
Debemos filtrar por edad y condición de hijo, y luego dentro de cada hogar calcular la edad del hijo menor:
aportes_ing <- eph_ind %>% select(codusu, nro_hogar, ch03, ch06) %>% filter(ch03==3 & ch06 %in% (0:17)) %>% #hijos menores group_by(codusu, nro_hogar) %>% mutate(edad_min = min(ch06))Group by() + Mutate() + Summarise()
Una vez calculada la edad del mas pequeño colapsamos la base de datos a un valor resumen. Para cada edad del hijo mas joven, calculamos la cantidad de hogares
aportes_ing <- eph_ind %>% select(codusu, nro_hogar, ch03, ch06) %>% filter(ch03==3 & ch06 %in% (0:17)) %>% #hijos menores group_by(codusu, nro_hogar) %>% mutate(edad_min = min(ch06)) %>% group_by(edad_min) %>% summarise(frecuency = n())Group by() + Mutate() + Summarise()
Nuevamente, es bueno ir desmenuzando cada paso para visualizar lo que ocurre
Group by() + Mutate() + Summarise()
Nuevamente, es bueno ir desmenuzando cada paso para visualizar lo que ocurre Para ello podemos ir segmentando el código cada parte, una vez que comprendemos qué ocurre y cómo es el output, agregamos un paso nuevo
Group by() + Mutate() + Summarise()
Nuevamente, es bueno ir desmenuzando cada paso para visualizar lo que ocurre Para ello podemos ir segmentando el código cada parte, una vez que comprendemos qué ocurre y cómo es el output, agregamos un paso nuevo
Y así sucesivamente, hasta entender el pipe completo de operaciones
Group by() + Mutate() + Summarise()
Nuevamente, es bueno ir desmenuzando cada paso para visualizar lo que ocurre Para ello podemos ir segmentando el código cada parte, una vez que comprendemos qué ocurre y cómo es el output, agregamos un paso nuevo
Y así sucesivamente, hasta entender el pipe completo de operaciones
En la slide siguiente hacemos esto mismo
Estos pasos ya deben empezar a resultar familiares para el lector
aportes_ing <- eph_ind %>% select(codusu, nro_hogar, ch03, ch06) %>% filter(ch03==3 & ch06 %in% (0:17))| codusu | nro_hogar | ch03 | ch06 |
|---|---|---|---|
| TQRMNOSWVHKMKOCDEHPJB00790947 | 1 | 3 | 12 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 1 | 3 | 7 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 1 | 3 | 15 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 1 | 3 | 11 |
| TQRMNOPXUHLMKUCDEGOIH00706727 | 1 | 3 | 10 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 1 | 3 | 7 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 1 | 3 | 9 |
| TQRMNOSUTHJMKTCDEHIBB00784269 | 1 | 3 | 11 |
| TQRMNOPPQHJLONCDEIIAD00785511 | 1 | 3 | 8 |
| TQRMNORQTHLOSNCDEIJAH00711164 | 1 | 3 | 4 |
| TQRMNORQTHLOSNCDEIJAH00711164 | 1 | 3 | 14 |
| TQRMNORQTHLOSNCDEIJAH00711164 | 1 | 3 | 6 |
| TQRMNOUPYHKOLQCDEGNFJ00789613 | 1 | 3 | 8 |
| TQRMNOSTPHLMLLCDEGKDB00705046 | 1 | 3 | 11 |
| TQRMNORSUHKOKQCDEFLID00787877 | 1 | 3 | 9 |
| TQRMNORSUHKOKQCDEFLID00787877 | 1 | 3 | 8 |
| TQRMNORSUHKOKQCDEFLID00787877 | 1 | 3 | 6 |
| TQRMNORSUHKOKQCDEFLID00787877 | 1 | 3 | 4 |
| TQRMNOQQWHKOKRCDEGOIH00789766 | 1 | 3 | 5 |
| TQRMNORRVHMOKRCDEOJAH00713918 | 1 | 3 | 3 |
| TQRMNOQWWHKOKPCDEGLDF00789500 | 1 | 3 | 2 |
| TQRMNOQYUHMOQRCDEIJAH00719559 | 1 | 3 | 2 |
| TQRMNOQWWHMMLQCDEIMBF00708621 | 1 | 3 | 5 |
| TQRMNOSSRHKMLSCDEFKID00787731 | 1 | 3 | 11 |
| TQRMNOSSRHKMLSCDEFKID00787731 | 1 | 3 | 7 |
| TQSMNOQXYHKMKTCDEHJGH00790304 | 1 | 3 | 6 |
| TQSMNOQXYHKMKTCDEHJGH00790304 | 1 | 3 | 2 |
| TQRMNOSUUHMOQQCDEIJAH00711284 | 1 | 3 | 14 |
| TQTMNOQXYHKMKTCDEHJGH00790305 | 1 | 3 | 13 |
| TQTMNOQXYHKMKTCDEHJGH00790305 | 1 | 3 | 11 |
| TQRMNORRXHLMLNCDEHJGH00706017 | 1 | 3 | 17 |
| TQRMNORRXHLMLNCDEHJGH00706017 | 1 | 3 | 11 |
| TQRMNOSQTHJMLNCDEHJGH00784389 | 1 | 3 | 2 |
| TQRMNQRPRHJOKUCDEHIBB00784312 | 1 | 3 | 11 |
| TQRMNQRPRHJOKUCDEHIBB00784312 | 1 | 3 | 9 |
| TQRMNQRPRHJOKUCDEHIBB00784312 | 1 | 3 | 2 |
| TQSMNOPUXHKOKPCDEGLDF00789498 | 1 | 3 | 12 |
| TQSMNOPUXHKOKPCDEGLDF00789498 | 1 | 3 | 3 |
| TQRMNORURHKNLNCDEHLEH00790435 | 1 | 3 | 9 |
| TQRMNORURHKNLNCDEHLEH00790435 | 1 | 3 | 8 |
| TQRMNORPTHKLKTCDEGLDF00789499 | 1 | 3 | 15 |
| TQRMNORPTHKLKTCDEGLDF00789499 | 1 | 3 | 12 |
| TQRMNORWRHKMKOCDEHPJB00790946 | 1 | 3 | 2 |
| TQRMNOSVWHLKMOCDEIIAD00785577 | 1 | 3 | 10 |
| TQRMNOQTYHLOKSCDEGOIH00707817 | 1 | 3 | 10 |
| TQRMNOPSRHJNNUCDEIIAD00785690 | 1 | 3 | 8 |
| TQRMNOPTVHMOKNCDEHMHF00711899 | 1 | 3 | 17 |
| TQRMNOPTVHMOKNCDEHMHF00711899 | 1 | 3 | 10 |
| TQRMNOPTVHMOKNCDEHMHF00711899 | 1 | 3 | 10 |
| TQRMNOPTVHMOKNCDEHMHF00711899 | 1 | 3 | 6 |
(a modo ilustrativo solo las primeras 50 filas)
Agrupamos a nivel hogares, con el identificador único que forman la variable codusu y nro_hogar y agregamos una columna nueva que calcula la edad mínima entre los hijos
aportes_ing <- eph_ind %>% select(codusu, nro_hogar, ch03, ch06) %>% filter(ch03==3 & ch06 %in% (0:17)) %>% #hijos menores group_by(codusu, nro_hogar) %>% mutate(edad_min = min(ch06))| codusu | nro_hogar | ch03 | ch06 | edad_min |
|---|---|---|---|---|
| TQRMNOSWVHKMKOCDEHPJB00790947 | 1 | 3 | 12 | 7 |
| TQRMNOSWVHKMKOCDEHPJB00790947 | 1 | 3 | 7 | 7 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 1 | 3 | 15 | 11 |
| TQRMNORRTHMMLLCDEOHCH00710640 | 1 | 3 | 11 | 11 |
| TQRMNOPXUHLMKUCDEGOIH00706727 | 1 | 3 | 10 | 10 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 1 | 3 | 7 | 7 |
| TQRMNOPPTHMMLMCDEGIBJ00712052 | 1 | 3 | 9 | 7 |
| TQRMNOSUTHJMKTCDEHIBB00784269 | 1 | 3 | 11 | 11 |
| TQRMNOPPQHJLONCDEIIAD00785511 | 1 | 3 | 8 | 8 |
| TQRMNORQTHLOSNCDEIJAH00711164 | 1 | 3 | 4 | 4 |
| TQRMNORQTHLOSNCDEIJAH00711164 | 1 | 3 | 14 | 4 |
| TQRMNORQTHLOSNCDEIJAH00711164 | 1 | 3 | 6 | 4 |
| TQRMNOUPYHKOLQCDEGNFJ00789613 | 1 | 3 | 8 | 8 |
| TQRMNOSTPHLMLLCDEGKDB00705046 | 1 | 3 | 11 | 11 |
| TQRMNORSUHKOKQCDEFLID00787877 | 1 | 3 | 9 | 4 |
| TQRMNORSUHKOKQCDEFLID00787877 | 1 | 3 | 8 | 4 |
| TQRMNORSUHKOKQCDEFLID00787877 | 1 | 3 | 6 | 4 |
| TQRMNORSUHKOKQCDEFLID00787877 | 1 | 3 | 4 | 4 |
| TQRMNOQQWHKOKRCDEGOIH00789766 | 1 | 3 | 5 | 5 |
| TQRMNORRVHMOKRCDEOJAH00713918 | 1 | 3 | 3 | 3 |
| TQRMNOQWWHKOKPCDEGLDF00789500 | 1 | 3 | 2 | 2 |
| TQRMNOQYUHMOQRCDEIJAH00719559 | 1 | 3 | 2 | 2 |
| TQRMNOQWWHMMLQCDEIMBF00708621 | 1 | 3 | 5 | 5 |
| TQRMNOSSRHKMLSCDEFKID00787731 | 1 | 3 | 11 | 7 |
| TQRMNOSSRHKMLSCDEFKID00787731 | 1 | 3 | 7 | 7 |
| TQSMNOQXYHKMKTCDEHJGH00790304 | 1 | 3 | 6 | 2 |
| TQSMNOQXYHKMKTCDEHJGH00790304 | 1 | 3 | 2 | 2 |
| TQRMNOSUUHMOQQCDEIJAH00711284 | 1 | 3 | 14 | 14 |
| TQTMNOQXYHKMKTCDEHJGH00790305 | 1 | 3 | 13 | 11 |
| TQTMNOQXYHKMKTCDEHJGH00790305 | 1 | 3 | 11 | 11 |
| TQRMNORRXHLMLNCDEHJGH00706017 | 1 | 3 | 17 | 11 |
| TQRMNORRXHLMLNCDEHJGH00706017 | 1 | 3 | 11 | 11 |
| TQRMNOSQTHJMLNCDEHJGH00784389 | 1 | 3 | 2 | 2 |
| TQRMNQRPRHJOKUCDEHIBB00784312 | 1 | 3 | 11 | 2 |
| TQRMNQRPRHJOKUCDEHIBB00784312 | 1 | 3 | 9 | 2 |
| TQRMNQRPRHJOKUCDEHIBB00784312 | 1 | 3 | 2 | 2 |
| TQSMNOPUXHKOKPCDEGLDF00789498 | 1 | 3 | 12 | 3 |
| TQSMNOPUXHKOKPCDEGLDF00789498 | 1 | 3 | 3 | 3 |
| TQRMNORURHKNLNCDEHLEH00790435 | 1 | 3 | 9 | 8 |
| TQRMNORURHKNLNCDEHLEH00790435 | 1 | 3 | 8 | 8 |
| TQRMNORPTHKLKTCDEGLDF00789499 | 1 | 3 | 15 | 12 |
| TQRMNORPTHKLKTCDEGLDF00789499 | 1 | 3 | 12 | 12 |
| TQRMNORWRHKMKOCDEHPJB00790946 | 1 | 3 | 2 | 2 |
| TQRMNOSVWHLKMOCDEIIAD00785577 | 1 | 3 | 10 | 10 |
| TQRMNOQTYHLOKSCDEGOIH00707817 | 1 | 3 | 10 | 10 |
| TQRMNOPSRHJNNUCDEIIAD00785690 | 1 | 3 | 8 | 8 |
| TQRMNOPTVHMOKNCDEHMHF00711899 | 1 | 3 | 17 | 6 |
| TQRMNOPTVHMOKNCDEHMHF00711899 | 1 | 3 | 10 | 6 |
| TQRMNOPTVHMOKNCDEHMHF00711899 | 1 | 3 | 10 | 6 |
| TQRMNOPTVHMOKNCDEHMHF00711899 | 1 | 3 | 6 | 6 |
Ahora agrupamos en base a esa columna nueva, es decir por cada valor posible de edad del hijo mas pequeño, y colapsamos la base a una medida resumen (frecuencia).
aportes_ing <- eph_ind %>% select(codusu, nro_hogar, ch03, ch06) %>% filter(ch03==3 & ch06 %in% (0:17)) %>% #hijos menores group_by(codusu, nro_hogar) %>% mutate(edad_min = min(ch06)) %>% group_by(edad_min) %>% summarise(frecuency = n())| edad_min | frecuency |
|---|---|
| 1 | 721 |
| 2 | 765 |
| 3 | 809 |
| 4 | 799 |
| 5 | 682 |
| 6 | 727 |
| 7 | 663 |
| 8 | 646 |
| 9 | 627 |
| 10 | 524 |
| 11 | 505 |
| 12 | 498 |
| 13 | 410 |
| 14 | 391 |
| 15 | 347 |
| 16 | 293 |
| 17 | 294 |
filter()select()rename()mutate()summarise()group_by()distinct()arrange()
Distinct()
Esta función nos permite quedarnos con observaciones únicas, es decir remover los duplicados
Distinct()
Esta función nos permite quedarnos con observaciones únicas, es decir remover los duplicados
Repasemos el ejercicio anterior, nos quedamos con hijos menores en cada hogar, calculamos la edad del menor y luego la frecuencia con que se da ese valor
Distinct()
Esta función nos permite quedarnos con observaciones únicas, es decir remover los duplicados
Repasemos el ejercicio anterior, nos quedamos con hijos menores en cada hogar, calculamos la edad del menor y luego la frecuencia con que se da ese valor
Al filtrar nos quedamos potencialmente con varios hijos en un mismo hogar que se duplican en la suma.
Distinct()
Esta función nos permite quedarnos con observaciones únicas, es decir remover los duplicados
Repasemos el ejercicio anterior, nos quedamos con hijos menores en cada hogar, calculamos la edad del menor y luego la frecuencia con que se da ese valor
Al filtrar nos quedamos potencialmente con varios hijos en un mismo hogar que se duplican en la suma. Lo que deberíamos hacer es quedarnos con una única observación por hogar
aportes_ing <- eph_ind %>% group_by(codusu, nro_hogar) %>% filter(ch03==3 & ch06 %in% (0:17)) %>% #hijos menores mutate(edad_min = min(ch06)) %>% distinct(codusu, nro_hogar, .keep_all = TRUE) %>% group_by(edad_min) %>% summarise(frecuency = n())Distinct()
Esta función nos permite quedarnos con observaciones únicas, es decir remover los duplicados
Repasemos el ejercicio anterior, nos quedamos con hijos menores en cada hogar, calculamos la edad del menor y luego la frecuencia con que se da ese valor
Al filtrar nos quedamos potencialmente con varios hijos en un mismo hogar que se duplican en la suma. Lo que deberíamos hacer es quedarnos con una única observación por hogar
aportes_ing <- eph_ind %>% group_by(codusu, nro_hogar) %>% filter(ch03==3 & ch06 %in% (0:17)) %>% #hijos menores mutate(edad_min = min(ch06)) %>% distinct(codusu, nro_hogar, .keep_all = TRUE) %>% group_by(edad_min) %>% summarise(frecuency = n())el argumento .keep_all = TRUE indica que además de las columnas que identifican a los duplicados, mantener el resto
Ahora la cantidad de hogares cambia en nuestro data frame resultante, respecto de la tabla anterior donde no consideramos el distintc()
| edad_min | frecuency |
|---|---|
| 1 | 330 |
| 2 | 370 |
| 3 | 382 |
| 4 | 393 |
| 5 | 345 |
| 6 | 359 |
| 7 | 350 |
| 8 | 349 |
| 9 | 338 |
| 10 | 307 |
| 11 | 311 |
| 12 | 314 |
| 13 | 286 |
| 14 | 298 |
| 15 | 291 |
| 16 | 284 |
| 17 | 288 |
filter()select()rename()mutate()summarise()group_by()distinct()arrange()
Arrange()
Arrange es la última función que veremos, esta nos permite ordenar de forma ascendente o descendente las observaciones
Por ejemplo, si dentro del hogar quisiéramos ordenar a los miembros por su edad
miembros <- eph_ind %>% group_by(codusu, nro_hogar) %>% arrange(ch06, .by_group = TRUE)miembros[1:10, c("codusu", "nro_hogar", "ch06")]## # A tibble: 10 x 3## # Groups: codusu, nro_hogar [5]## codusu nro_hogar ch06## <chr> <int> <int>## 1 TQRMNOPPQHJKKSCDEHJGH00790191 1 83## 2 TQRMNOPPQHJKLLCDEGOIH00783953 1 12## 3 TQRMNOPPQHJKLLCDEGOIH00783953 1 56## 4 TQRMNOPPQHJKTMCDEIJAH00786549 1 18## 5 TQRMNOPPQHJKTMCDEIJAH00786549 1 42## 6 TQRMNOPPQHJKTMCDEIJAH00786549 1 50## 7 TQRMNOPPQHJLONCDEIIAD00785511 1 8## 8 TQRMNOPPQHJLONCDEIIAD00785511 1 41## 9 TQRMNOPPQHJLONCDEIIAD00785511 1 44## 10 TQRMNOPPQHJMKTCDEGGFB00783084 1 8Arrange()
O en nuestro ejemplo anterior, si quisiéramos ordenar las edades de los hijos menores de mayor a menor en vez de al revés
aportes_ing <- aportes_ing %>% arrange(desc(edad_min))| edad_min | frecuency |
|---|---|
| 17 | 288 |
| 16 | 284 |
| 15 | 291 |
| 14 | 298 |
| 13 | 286 |
| 12 | 314 |
| 11 | 311 |
| 10 | 307 |
| 9 | 338 |
| 8 | 349 |
| 7 | 350 |
| 6 | 359 |
| 5 | 345 |
| 4 | 393 |
| 3 | 382 |
| 2 | 370 |
| 1 | 330 |
EL ABC de dplyr
BIEN!
EL ABC de dplyr
BIEN!
Hemos recorrido las funciones principales de dplyr. Ahora ya estamos equipados para comenzar a transformar datos y manipularlos.
EL ABC de dplyr
BIEN!
Hemos recorrido las funciones principales de dplyr. Ahora ya estamos equipados para comenzar a transformar datos y manipularlos.
Antes de terminar, a modo de extra, introduciremos algunos conceptos adicionales, que permiten fácilmente escalar y replicar algunas de estas operaciones de manera rápida
EL ABC de dplyr
BIEN!
Hemos recorrido las funciones principales de dplyr. Ahora ya estamos equipados para comenzar a transformar datos y manipularlos.
Antes de terminar, a modo de extra, introduciremos algunos conceptos adicionales, que permiten fácilmente escalar y replicar algunas de estas operaciones de manera rápida
Con un esfuerzo marginal, podremos aplicar nuestro ABC de manera eficiente, corriendo una sola linea para transformar múltiples variables a la vez.
EL ABC de dplyr
BIEN!
Hemos recorrido las funciones principales de dplyr. Ahora ya estamos equipados para comenzar a transformar datos y manipularlos.
Antes de terminar, a modo de extra, introduciremos algunos conceptos adicionales, que permiten fácilmente escalar y replicar algunas de estas operaciones de manera rápida
Con un esfuerzo marginal, podremos aplicar nuestro ABC de manera eficiente, corriendo una sola linea para transformar múltiples variables a la vez.
Veamos algunos ejemplos simples para despertar la curiosidad sobre este punto
Operaciones para múltiples columnas
A veces es necesario realizar una misma operación sobre varias columnas a la vez
Operaciones para múltiples columnas
A veces es necesario realizar una misma operación sobre varias columnas a la vez
Antes vimos la función rename_with() que nos permitía renombrar muchas columnas al mismo tiempo. Veamos cómo funciona esta lógica para mutate() y summarise()
Operaciones para múltiples columnas
A veces es necesario realizar una misma operación sobre varias columnas a la vez
Antes vimos la función rename_with() que nos permitía renombrar muchas columnas al mismo tiempo. Veamos cómo funciona esta lógica para mutate() y summarise()
Por ejemplo, si en la eph quisiéramos pasar a dolares todas las columnas de ingreso, la forma rudimentaria de hacerlo sería la siguiente
eph_ind_usd <- eph_ind %>% mutate(p21_usd = p21/390, tot_p12_usd = tot_p12/390, t_vi_usd = t_vi/390, p47t_usd = p47t/390, itf_usd = itf/390) %>% select(ends_with("USD")) #solo muestro columnas nuevasBueno... es un poco tedioso pero lo podemos hacer. Ahora imagínense si estas fueran mas columnas, muchas mas, hacerlo a mano sería pesado.
Across()
Para eso existe la función across() que nos permite reducir el trabajo de código y lograr el mismo resultado.
Across()
Para eso existe la función across() que nos permite reducir el trabajo de código y lograr el mismo resultado. Veamos qué argumentos nos demanda esta función
across(.cols, .fns, ..., .names = NULL)
Across()
Para eso existe la función across() que nos permite reducir el trabajo de código y lograr el mismo resultado. Veamos qué argumentos nos demanda esta función
across(.cols, .fns, ..., .names = NULL)
- cuáles columnas modificar:
.cols - qué función aplicar:
.fn - opcionalmente especificar nombres nuevos
.names
Across()
Para eso existe la función across() que nos permite reducir el trabajo de código y lograr el mismo resultado. Veamos qué argumentos nos demanda esta función
across(.cols, .fns, ..., .names = NULL)
- cuáles columnas modificar:
.cols - qué función aplicar:
.fn - opcionalmente especificar nombres nuevos
.names
Probemos con el caso anterior
eph_ind_usd <- eph_ind %>% mutate(across(.cols=c(p21,tot_p12, t_vi, p47t, itf), .fns = function(x) (x/390), .names = "{col}_USD")) %>% select(ends_with("_USD")) #solo muestro columnas nuevasAcross()
Para eso existe la función across() que nos permite reducir el trabajo de código y lograr el mismo resultado. Veamos qué argumentos nos demanda esta función
across(.cols, .fns, ..., .names = NULL)
- cuáles columnas modificar:
.cols - qué función aplicar:
.fn - opcionalmente especificar nombres nuevos
.names
Probemos con el caso anterior
eph_ind_usd <- eph_ind %>% mutate(across(.cols=c(p21,tot_p12, t_vi, p47t, itf), .fns = function(x) (x/390), .names = "{col}_USD")) %>% select(ends_with("_USD")) #solo muestro columnas nuevasnames = "{col}_USD crea para un nombre para esas columnas transformadas, que sea el mismo nombre de la columna original mas el sufijo "_USD"
head(eph_ind_usd, 10)## ipcf_usd p21_USD tot_p12_USD t_vi_USD p47t_USD itf_USD## 1 117.9487 51.28205 0.00000 66.66666667 117.94871795 117.9487## 2 410.7692 0.00000 0.00000 113.84615385 113.84615385 821.5385## 3 410.7692 0.00000 0.00000 707.69230769 707.69230769 821.5385## 4 0.0000 0.00000 0.00000 -0.02307692 -0.02307692 0.0000## 5 0.0000 0.00000 0.00000 -0.02307692 -0.02307692 0.0000## 6 26.5641 0.00000 0.00000 12.82051282 12.82051282 132.8205## 7 26.5641 0.00000 38.46154 0.00000000 38.46153846 132.8205## 8 26.5641 0.00000 0.00000 0.00000000 0.00000000 132.8205## 9 26.5641 0.00000 0.00000 81.53846154 81.53846154 132.8205## 10 26.5641 0.00000 0.00000 0.00000000 0.00000000 132.8205Across()
La expresión que le pasemos al argumento .fns puede resultar un poco rara
Across()
La expresión que le pasemos al argumento .fns puede resultar un poco rara
Tratemos de entender qué le estamos diciendo a R con function(x) x/390
Across()
La expresión que le pasemos al argumento .fns puede resultar un poco rara
Tratemos de entender qué le estamos diciendo a R con function(x) x/390
function(x)indica que vamos a pasar una operación para que aplique sobre x
Across()
La expresión que le pasemos al argumento .fns puede resultar un poco rara
Tratemos de entender qué le estamos diciendo a R con function(x) x/390
function(x)indica que vamos a pasar una operación para que aplique sobre xx/390es esa operación
Across()
La expresión que le pasemos al argumento .fns puede resultar un poco rara
Tratemos de entender qué le estamos diciendo a R con function(x) x/390
function(x)indica que vamos a pasar una operación para que aplique sobre xx/390es esa operación
¿Y qué valor va a tomar x?...
Across()
La expresión que le pasemos al argumento .fns puede resultar un poco rara
Tratemos de entender qué le estamos diciendo a R con function(x) x/390
function(x)indica que vamos a pasar una operación para que aplique sobre xx/390es esa operación
¿Y qué valor va a tomar x?...
x será cada una de las columnas que indicamos antes
Across()
La expresión que le pasemos al argumento .fns puede resultar un poco rara
Tratemos de entender qué le estamos diciendo a R con function(x) x/390
function(x)indica que vamos a pasar una operación para que aplique sobre xx/390es esa operación
¿Y qué valor va a tomar x?...
x será cada una de las columnas que indicamos antes , es decir esa operación se aplica sobre los valores que tome cada una de estas columna en cada observación
Across()
Veamos otro ejemplo, supongamos que por aglomerado queremos expresar los datos de ingreso como porcentaje del promedio.
eph_ind_mean <- eph_ind %>% group_by(aglomerado) %>% mutate(across(.cols = c(p21, tot_p12, t_vi, p47t, itf), .fns= function(x) x/mean(x, na.rm = TRUE), .names = "{col}_percent")) %>% select(aglomerado, ends_with("_percent"))Across()
Otro ejemplo, si quisiéramos redondear a dos decimales todas las columnas que sean numéricas
Across()
Otro ejemplo, si quisiéramos redondear a dos decimales todas las columnas que sean numéricas
De la misma familia que contains(), ends_with(), star_with() existe where() que aplica la función a todas las columnas que satisfagan una condición lógica.
Across()
Otro ejemplo, si quisiéramos redondear a dos decimales todas las columnas que sean numéricas
De la misma familia que contains(), ends_with(), star_with() existe where() que aplica la función a todas las columnas que satisfagan una condición lógica.
En este caso, esa condición es que sea numérica
eph_ind_r <- eph_ind %>% mutate(across(where(is.numeric), .fns= function(x) round(x, digits=2)))Notar que en este caso estaremos pisando (reemplazando) las columnas originales
Across()
También podemos aplicarlo con summarise()
Si quisiéramos para una lista de columnas la media y el desvío de cada una, podemos escribir
eph_ind %>% summarise(across(.cols = c(p21, ipcf, itf), .fns = list(mean=mean, sd=sd), na.rm=TRUE))## p21_mean p21_sd ipcf_mean ipcf_sd itf_mean itf_sd## 1 25559.51 52545.68 37628.33 47253.11 119202.9 127728.3Notar que en este caso mediante la función list() paso una lista de funciones, para cada columna calculo tanto la media como el desvío
Extra: Donde consultar?
Material recomendado para revisar y extender estos contenidos:
Para extender la noción y explicación de los temas vistos, así como encontrar ejemplos y aplicaciones con otras bases de datos revisar el libro de consulta de base. Se recomienda este material para introducirse en cada tema, lograr una comprensión conceptual y poder practicar con ejemplos concretos.
Extra: Donde consultar?
Material recomendado para revisar y extender estos contenidos:
Para extender la noción y explicación de los temas vistos, así como encontrar ejemplos y aplicaciones con otras bases de datos revisar el libro de consulta de base. Se recomienda este material para introducirse en cada tema, lograr una comprensión conceptual y poder practicar con ejemplos concretos.
- Cheat-Sheets: ayuda memoria y resumen
Este material funciona como ayuda memoria o "machete". En dos hojas agrupa las funciones por tema, y explica brevemente para qué sirven y qué argumentos requiere cada una. Recomendado para ganar agilidad al escribir códigos. Se listan solo algunos ejemplos, en la sección material extra encontrarán más.
Extra: Identación, estilo y orden
Veamos algunas buenas prácticas y conjunto de recomendaciones a la hora de escribir código.
Extra: Identación, estilo y orden
Veamos algunas buenas prácticas y conjunto de recomendaciones a la hora de escribir código.
Para facilitar la lectura y el orden de los códigos una buena práctica es mantener una identificación, es decir un espacio respecto del margen.
Extra: Identación, estilo y orden
Veamos algunas buenas prácticas y conjunto de recomendaciones a la hora de escribir código.
Para facilitar la lectura y el orden de los códigos una buena práctica es mantener una identificación, es decir un espacio respecto del margen.
Esto ayuda a identificar rápidamente el objeto inicial, las distintas operaciones que se aplican y la secuencialidad
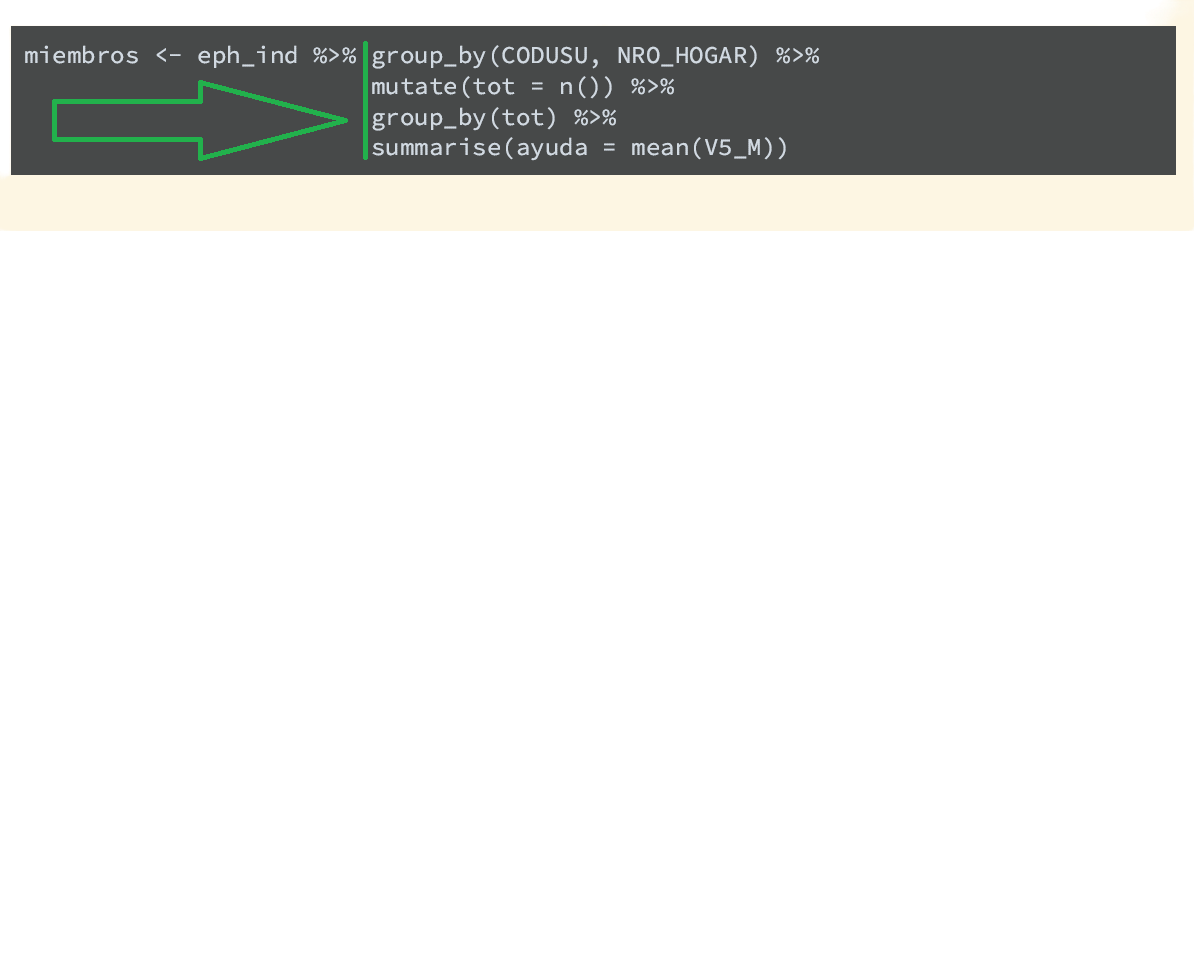
Extra: Identación estilo y orden
Mismo aplica por ejemplo para especificar argumentos de una función, sobre todo cuando estos son largos
long_function <- function(a = "a long argument", b = "another argument", c = "another long argument") {}Extra: Identación estilo y orden
Mismo aplica por ejemplo para especificar argumentos de una función, sobre todo cuando estos son largos
long_function <- function(a = "a long argument", b = "another argument", c = "another long argument") {}Si les interesa leer más sobre esto ver el siguiente link
Para un resumen de las recomendaciones generales ver en la bibliografía base
Extra: Identación estilo y orden
De esas recomendaciones generales aquí haremos énfasis en dos, evitar mayúsculas y espacios
#Procurarsubset <- eph_ind %>% filter(!is.na(IPCF)) %>% #EvitarSUBSET <- eph_ind %>% filter(!is.na(IPCF)) %>% )Extra: Identación estilo y orden
De esas recomendaciones generales aquí haremos énfasis en dos, evitar mayúsculas y espacios
#Procurarsubset <- eph_ind %>% filter(!is.na(IPCF)) %>% #EvitarSUBSET <- eph_ind %>% filter(!is.na(IPCF)) %>% )#Así SÍmean(x, na.rm = TRUE)#Así NOmean (x ,na.rm=TRUE)Extra: Identación estilo y orden
Otra buena práctica, es documentar y comentar sus códigos
Extra: Identación estilo y orden
Otra buena práctica, es documentar y comentar sus códigos
Esto es muy útil para compartir los códigos con otros pero sobre todo para su "yo del futuro"
#Calculo diferencias en horas y en ingreso por urbano-ruralgender_diff_ur <- subset %>% mutate(URBANO = ifelse(mas_500=="S", 1, 0)) %>% group_by(sexo, urbano) %>% summarise(hs = mean(pp3e_tot, na.rm=TRUE), #uso media ipcf = median(ipcf, na.rm = TRUE)) #uso medianaExtra: Identación estilo y orden
Otra buena práctica, es documentar y comentar sus códigos
Esto es muy útil para compartir los códigos con otros pero sobre todo para su "yo del futuro"
#Calculo diferencias en horas y en ingreso por urbano-ruralgender_diff_ur <- subset %>% mutate(URBANO = ifelse(mas_500=="S", 1, 0)) %>% group_by(sexo, urbano) %>% summarise(hs = mean(pp3e_tot, na.rm=TRUE), #uso media ipcf = median(ipcf, na.rm = TRUE)) #uso medianaTampoco hace falta que sea exagerado, es una ayuda para entender el código, no un detalle pormenorizado.
Extra: Identación estilo y orden
Por último, es fundamental ordenar el script, asignándole una estructura. Para esto:
#----
Extra: Identación estilo y orden
Por último, es fundamental ordenar el script, asignándole una estructura. Para esto:
#----
Anteponiéndolo a la palabra o frase, la cantidad de # que usemos indica la jerarquía
Para que se incorpore al índice necesitamos indicar al menos 4 guiones seguidos: ----
Extra: Identación estilo y orden
Por último, es fundamental ordenar el script, asignándole una estructura. Para esto:
#----
Anteponiéndolo a la palabra o frase, la cantidad de # que usemos indica la jerarquía
Para que se incorpore al índice necesitamos indicar al menos 4 guiones seguidos: ----
En las próximas clases veremos cómo setear el path y navegar por las diferentes subcarpetas
En las próximas clases veremos cómo setear el path y navegar por las diferentes subcarpetas
En los trabajos prácticos se espera que escriban sus códigos con estas buenas prácticas
En las próximas clases veremos cómo setear el path y navegar por las diferentes subcarpetas
En los trabajos prácticos se espera que escriban sus códigos con estas buenas prácticas